贴标签机多边形图像转换为标签阻塞语义分割图像
这个例子展示了如何将存储在一个多边形标签groundTruth对象到一个标签阻塞语义分割图像适合工作流。
您可以使用图像贴标签机应用在计算机视觉工具箱标签图片太大,适合记忆和多分辨率图像。有关更多信息,请参见标签图片标志大图像(计算机视觉工具箱)。图片标志应用程序不支持图像像素标签阻塞。万博1manbetx你只能使用ROI创建标签形状如多边形、矩形、和线条。这个例子展示了如何使用polyToBlockedImage函数将多边形roi转化为一个pixel-labeled阻塞图像语义分割工作流。
创建一个阻塞图像使用修改后的版本的训练图像包含肿瘤的淋巴结组织,从CAMELYON16数据集。修改后的图像有三个粗分辨率的水平。空间引用已调整执行一致的长宽比和注册功能在每个级别。
bim = blockedImage (“tumor_091R.tif”);
地面实况数据加载标签
这个示例加载presavedgroundTruth对象,gTruth.mat由标签阻塞图像数据荡妇使用图像贴标签机应用。groundTruth对象存储多边形标签显示在图中。正常组织的ROI中概述绿色,肿瘤组织ROI是用红色标出。您可以导出自己的图片标志贴上地面实况数据应用选择出口然后到工作空间。变量的名字gTruth。
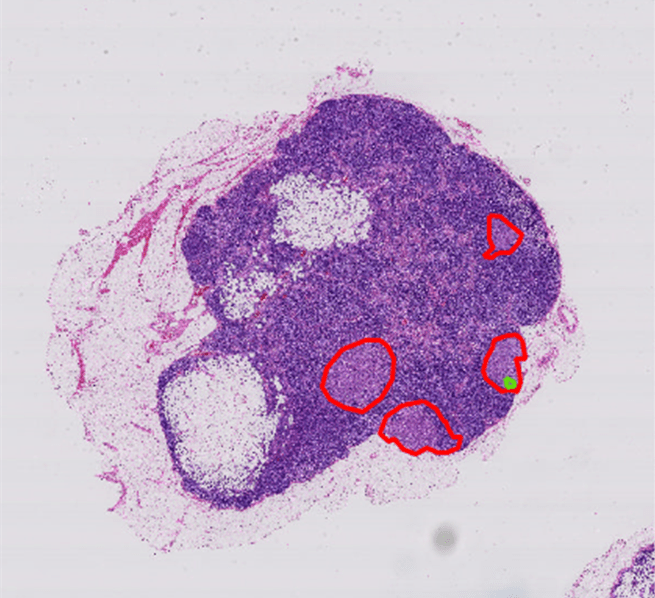
负载gTruth.mat
提取ROI位置和标签数据
的LabelData财产的gTruth对象存储多边形标签数据为每个标签包含一个列的表。
labelData = gTruth.LabelData
labelData =1×2表正常的肿瘤属于__________{4×2双}{4×1细胞}
使用辅助函数gTruthtoXY,在这个例子中,定义转换xy -位置数据和存储在标签labelData接受格式作为输入polyToBlockedImage函数。gTruthtoXY分配的数字标签1正常组织的ROI和标签2肿瘤组织roi。
[roiPositions, roiLabels] = gTruthtoXY (labelData)
roiPositions =5×1单元阵列{4×2双}{14×2双}{16×2双}{20×2双}{12×2双}
roiLabels =5×11 2 2 2 2
创建标记阻止了图像
选择所需的分辨率水平为新的阻塞的形象。这个选择是一个权衡效率和准确性。使用粗分辨率水平减少处理时间和存储大小。使用一个更精细的分辨率水平增加了保存在面具的详细程度。您可以使用粗分辨率水平定期roi,像多边形。小,徒手画的roi,高分辨率的水平是更合适的。对于这个示例,使用一个中等分辨率水平。
maskLevel = 2;
指定图像大小为新图像与原始图像的阻塞,荡妇在所需的分辨率水平。
图象尺寸= bim.Size (maskLevel 1:2);
创建一个标记了的形象。保持最初的封锁的空间参考图像,荡妇在所需的分辨率水平。默认情况下,polyToBlockedImage指定像素不落在任何ROI数字标签0。
bLabeled = polyToBlockedImage (roiPositions roiLabels图象尺寸,…BlockSize = bim.BlockSize (maskLevel 1:2),…WorldStart = bim.WorldStart (maskLevel 1:2), WorldEnd = bim.WorldEnd (maskLevel 1:2));
显示标签阻塞图像叠加在原始图像上。相对应的区域肿瘤和正常组织的多边形groundTruth分别为对象所示红色和绿色。区分正常组织以外的roi从背景中使用二进制面具,看到的创建标记阻止图像roi和面具。
hbim = bigimageshow (bim);showlabels (hbim bLabeled Colormap = [0 0 1;0 1 0;1 0 0])

万博1manbetx支持函数
的gTruthtoXYhelper函数转换多边形ROI坐标和标签数据存储在表中labelData成细胞阵列适合输入polyToBlockedImage函数。
函数[roiPositions, roiLabels] = gTruthtoXY (labelData) totalROIs =元素个数(labelData{1 1}) +元素个数(labelData {1,2} {:});roiPositions =细胞(totalROIs, 1);roiLabels = 0 (totalROIs, 1);%获得labelData表的标签名称labelName = labelData.Properties.VariableNames;roiIdx = 1;%初始化ROI指数%遍历所有标签%分配一个数字标签2的肿瘤组织;1正常组织为j = 1:元素个数(labelData)%所有roi对于一个给定的标签data = labelData {1, j} {:};如果(isequal (labelName {j},“肿瘤”))为k = 1:元素个数(数据)roiLabels (roiIdx) = 2;roiPositions {roiIdx} = {k}数据;roiIdx = roiIdx + 1;结束其他的%为其他ROI标签roiLabels (roiIdx) = 1;roiPositions {roiIdx} =数据;roiIdx = roiIdx + 1;结束结束结束
另请参阅
polyToBlockedImage|blockedImage|图片标志(计算机视觉工具箱)
相关的话题
- 标签图片标志大图像(计算机视觉工具箱)
- 创建标记阻止图像roi和面具
