三维大脑肿瘤分割使用深度学习
这个例子展示了如何训练3 d U-Net神经网络和执行语义分割三维医学图像的脑瘤。
这个例子展示了如何训练3 d U-Net网络也提供了一个pretrained网络。如果您选择训练网络,使用CUDA-capable NVIDIA™GPU计算能力3.0或更高版本强烈推荐(需要并行计算工具箱™)。
介绍
语义分割包括标记图像的每个像素或体素的三维体积与一个类。
这个例子说明了深度学习方法的使用语义部分脑部肿瘤的磁共振成像(MRI)扫描。它还展示了如何执行二元分割,每个立体像素标记为肿瘤或背景。
医学图像分割的一个挑战是类失衡阻碍了培训在使用传统的交叉熵的数据损失。这个例子解决问题通过使用加权多级骰子损失函数[4]。重类有助于对抗骰子点数较大区域的影响,使网络更容易学会段较小的地区。
这个例子执行大脑肿瘤分割使用3 d U-Net架构[1]。U-Net是一个快速、高效和简单的网络,在语义细分领域中已变得很流行。
下载培训、验证和测试数据
下面的例子使用了小鬼数据集[2]。小鬼数据集包含了脑部肿瘤的MRI扫描,即神经胶质瘤,这是最常见的原发性脑恶性肿瘤。数据文件的大小~ 7 GB。如果你不想下载的后代的数据集,然后直接下载Pretrained网络和测试集样本在这个例子中部分。
创建一个目录来存储小鬼数据集。
imageDir = fullfile (tempdir,“小鬼”);如果~存在(imageDir“dir”mkdir (imageDir);结束
下载的后代数据,去医学分割Decathalon网站,点击“下载数据”链接。下载“Task01_BrainTumour。焦油”文件[3]。解压TAR文件到指定的目录imageDir变量。当成功解压缩,imageDir将包含一个目录命名Task01_BrainTumour这有三个子目录:imagesTr,imagesTs,labelsTr。
数据集包含750个4 - d卷,每个代表一个堆栈的3 d图像。每个4 d卷大小240 -了- 240 - 155 - 4,在前三个维度对应高度,宽度和深度的三维体积的形象。第四维对应于不同的扫描方式。每个图像体积都有对应的像素标签。484数据集分为训练卷和286测试卷。
预处理训练和验证数据
训练3 d U-Net网络更有效率,预处理MRI数据使用helper函数preprocessBraTSdataset。这个函数是附加到例子作为支持文件。万博1manbetx
helper函数执行这些操作:
农作物数据包含一个区域主要是大脑和肿瘤。裁剪数据减少了数据的大小,同时保留最关键的部分每个MRI体积及其相应的标签。
正常化每个独立形态的体积减去均值和除以标准差剪裁的大脑区域。
将数据集分为训练、验证和测试集。
预处理的数据可能需要30分钟才能完成。
sourceDataLoc = [imageDir filesep“Task01_BrainTumour”];preprocessDataLoc = fullfile (tempdir,“小鬼”,“preprocessedDataset”);preprocessBraTSdataset (preprocessDataLoc sourceDataLoc);
创建随机补丁提取数据存储的培训和验证
使用一块随机抽取数据存储提要训练数据网络和验证培训的进展。这个数据存储从地面实况图像和相应的像素提取随机补丁标签数据。修补是一种常见的技术来防止内存耗尽时训练和任意大卷
首先,将训练图像存储在一个imageDatastore。因为MAT-file格式是一个非标准的图像格式,您必须使用一个MAT-file读者使读取图像数据。您可以使用辅助MAT-file读者,matRead。这个函数是附加到例子作为支持文件。万博1manbetx
volReader = @ (x) matRead (x);volLoc = fullfile (preprocessDataLoc,“imagesTr”);volds = imageDatastore (volLoc,…“FileExtensions”,“.mat”,“ReadFcn”,volReader);
创建一个pixelLabelDatastore存储标签。
labelReader = @ (x) matRead (x);lblLoc = fullfile (preprocessDataLoc,“labelsTr”);一会= [“背景”,“肿瘤”];pixelLabelID = [0 1];pixelLabelID pxds = pixelLabelDatastore (lblLoc,一会,…“FileExtensions”,“.mat”,“ReadFcn”,labelReader);
预览一个图像体积和标签。显示标签卷使用labelvolshow函数。把背景设置可见性完全透明的背景标签(1)0。
体积=预览(volds);标签=预览(pxds);图h = labelvolshow(标签、卷(:,:,:1));h.LabelVisibility (1) = 0;
创建一个randomPatchExtractionDatastore从图像数据存储和数据存储像素标签。指定一个补丁的大小64 - - 64 - 64像素点。指定“PatchesPerImage”提取16从每组随机定位补丁的数量和标签在训练。指定一个mini-batch大小8。
patchSize = (64 64 64);patchPerImage = 16;miniBatchSize = 8;patchds = randomPatchExtractionDatastore (volds pxds patchSize,…“PatchesPerImage”,patchPerImage);patchds。MiniBatchSize = MiniBatchSize;
增加训练数据使用变换函数与自定义的helper函数指定的预处理操作augment3dPatch。的augment3dPatch函数随机旋转和反映了训练数据训练更健壮。这个函数是附加到例子作为支持文件。万博1manbetx
dsTrain =变换(patchds @augment3dPatch);
创建一个randomPatchExtrationDatastore包含验证数据。您可以使用验证数据来评估是否网络不断学习,随着时间的推移underfitting或过度拟合。
volLocVal = fullfile (preprocessDataLoc,“imagesVal”);voldsVal = imageDatastore (volLocVal,…“FileExtensions”,“.mat”,“ReadFcn”,volReader);lblLocVal = fullfile (preprocessDataLoc,“labelsVal”);pixelLabelID pxdsVal = pixelLabelDatastore (lblLocVal,一会,…“FileExtensions”,“.mat”,“ReadFcn”,labelReader);dsVal = randomPatchExtractionDatastore (voldsVal pxdsVal patchSize,…“PatchesPerImage”,patchPerImage);dsVal。MiniBatchSize = MiniBatchSize;
建立三维U-Net层
这个示例使用的一种变体3 d U-Net网络[1]。U-Net,最初的一系列卷积层点缀着麦克斯池层,先后降低输入图像的分辨率。这些层是紧随其后的是一系列的卷积层点缀着upsampling运营商,先后增加输入图像的分辨率。U-Net名称来自这样一个事实:网络可以用一个对称的形状像字母u这个示例修改U-Net用零填充曲线玲珑,这样输入和输出曲线玲珑有相同的大小。
这个例子定义了三维U-Net利用深度学习工具箱™的层,包括:
image3dInputLayer——3 d图像输入层convolution3dLayer——三维卷积为卷积神经网络层batchNormalizationLayer——批量标准化层reluLayer漏水的解决线性单元层maxPooling3dLayer——3 d max池层transposedConv3dLayer——转置三维卷积层softmaxLayer——Softmax输出层concatenationLayer——连接层
这个例子还定义了一个定制的骰子损失层,命名dicePixelClassification3dLayer,解决数据的类不平衡问题[4]。这一层是附加到例子作为支持文件。万博1manbetx有关更多信息,请参见自定义像素分类层与骰子的损失。
第一层,imageInput3dLayer作用于图像的大小64 - - 64 - 64像素点。
inputSize = (64 64 64 4);inputLayer = image3dInputLayer (inputSize,“归一化”,“没有”,“名字”,“输入”);
图像输入层的收缩路径3 d U-Net紧随其后。承包路径包括三个编码器模块。每个编码器包含两个卷积与3-by-3-by-3过滤层特征图的数量增加一倍,紧随其后的是一个非线性激活使用reLu层。第一次卷积也紧随其后的是一批标准化层。每个编码器以马克斯池层半每个维度的图像分辨率。
给所有的层独特的名字。层编码器的名称从“en”其次是编码器的索引模块。例如,“en1”表示第一个编码器模块。
numFiltersEncoder = [32 64;64 128;128 256);层= [inputLayer];为模块= 1:3 modtag = num2str(模块);encoderModule = [convolution3dLayer (numFiltersEncoder(模块,1),…“填充”,“相同”,“WeightsInitializer”,“narrow-normal”,…“名字”,(“en”modtag,“_conv1”]);batchNormalizationLayer (“名字”,(“en”modtag,“_bn”]);reluLayer (“名字”,(“en”modtag,“_relu1”]);convolution3dLayer (numFiltersEncoder(模块,2),…“填充”,“相同”,“WeightsInitializer”,“narrow-normal”,…“名字”,(“en”modtag,“_conv2”]);reluLayer (“名字”,(“en”modtag,“_relu2”]);maxPooling3dLayer (2“步”2,“填充”,“相同”,…“名字”,(“en”modtag,“_maxpool”]);];层=[层;encoderModule];结束
创建的扩展路径3 d U-Net。扩展路径由四个解码器模块。所有的解码器都含有两个卷积层与3-by-3-by-3过滤器特征图的数量减半,紧随其后的是一个非线性激活使用reLu层。前三个解码器的结论与转置卷积层upsamples图像的2倍。最后卷积译码器包括一个层,每个立体像素的特征向量映射到类。
给所有的层独特的名字。层译码器的名称从“德”其次是译码器的索引模块。例如,“de4”表示第四解码器模块。
numFiltersDecoder = (256 512;256 256;128 128;64 64);decoderModule4 = [convolution3dLayer (numFiltersDecoder (1, 1),…“填充”,“相同”,“WeightsInitializer”,“narrow-normal”,…“名字”,“de4_conv1”);reluLayer (“名字”,“de4_relu1”);convolution3dLayer (numFiltersDecoder (1、2),…“填充”,“相同”,“WeightsInitializer”,“narrow-normal”,…“名字”,“de4_conv2”);reluLayer (“名字”,“de4_relu2”);transposedConv3dLayer (numFiltersDecoder (1、2),“步”2,…“名字”,“de4_transconv”);];decoderModule3 = [convolution3dLayer (numFiltersDecoder (2, 1),…“填充”,“相同”,“WeightsInitializer”,“narrow-normal”,…。“名字”,“de3_conv1”);reluLayer (“名字”,“de3_relu1”);convolution3dLayer (numFiltersDecoder (2, 2),…“填充”,“相同”,“WeightsInitializer”,“narrow-normal”,…“名字”,“de3_conv2”);reluLayer (“名字”,“de3_relu2”);transposedConv3dLayer (numFiltersDecoder (2, 2),“步”2,…“名字”,“de3_transconv”);];decoderModule2 = [convolution3dLayer (numFiltersDecoder (3,1),…“填充”,“相同”,“WeightsInitializer”,“narrow-normal”,…“名字”,“de2_conv1”);reluLayer (“名字”,“de2_relu1”);convolution3dLayer (numFiltersDecoder (3, 2),…“填充”,“相同”,“WeightsInitializer”,“narrow-normal”,…“名字”,“de2_conv2”);reluLayer (“名字”,“de2_relu2”);transposedConv3dLayer (numFiltersDecoder (3, 2),“步”2,…“名字”,“de2_transconv”);];
最后卷积译码器包括一个层,每个立体像素的特征向量映射到每个两个类(肿瘤和背景)。自定义骰子像素分类层重量损失函数增加骰子上的小肿瘤区域的影响得分。
numLabels = 2;decoderModuleFinal = [convolution3dLayer (numFiltersDecoder (4,1),…“填充”,“相同”,“WeightsInitializer”,“narrow-normal”,…“名字”,“de1_conv1”);reluLayer (“名字”,“de1_relu1”);convolution3dLayer (numFiltersDecoder (4,2),…“填充”,“相同”,“WeightsInitializer”,“narrow-normal”,…“名字”,“de1_conv2”);reluLayer (“名字”,“de1_relu2”);numLabels convolution3dLayer (1,“名字”,“convLast”);softmaxLayer (“名字”,“softmax”);dicePixelClassification3dLayer (“输出”);];
连接输入层与第四解码器和编码器模块模块。其他解码器模块添加到图层图表作为独立的分支。
层=[层;decoderModule4];lgraph = layerGraph(层);lgraph = addLayers (lgraph decoderModule3);lgraph = addLayers (lgraph decoderModule2);lgraph = addLayers (lgraph decoderModuleFinal);

使用连接层连接第二reLu层转置卷积的每个编码器模块层同等大小的译码器模块。每个连接层的输出连接到第一个卷积译码器模块的层。
concat1 = concatenationLayer (4 2“名字”,“concat1”);lgraph = addLayers (lgraph concat1);lgraph = connectLayers (lgraph,“en1_relu2”,“concat1 /三机一体”);lgraph = connectLayers (lgraph,“de2_transconv”,“concat1 / in2”);lgraph = connectLayers (lgraph,“concat1 /出”,“de1_conv1”);concat2 = concatenationLayer (4 2“名字”,“concat2”);lgraph = addLayers (lgraph concat2);lgraph = connectLayers (lgraph,“en2_relu2”,“concat2 /三机一体”);lgraph = connectLayers (lgraph,“de3_transconv”,“concat2 / in2”);lgraph = connectLayers (lgraph,“concat2 /出”,“de2_conv1”);concat3 = concatenationLayer (4 2“名字”,“concat3”);lgraph = addLayers (lgraph concat3);lgraph = connectLayers (lgraph,“en3_relu2”,“concat3 /三机一体”);lgraph = connectLayers (lgraph,“de4_transconv”,“concat3 / in2”);lgraph = connectLayers (lgraph,“concat3 /出”,“de3_conv1”);
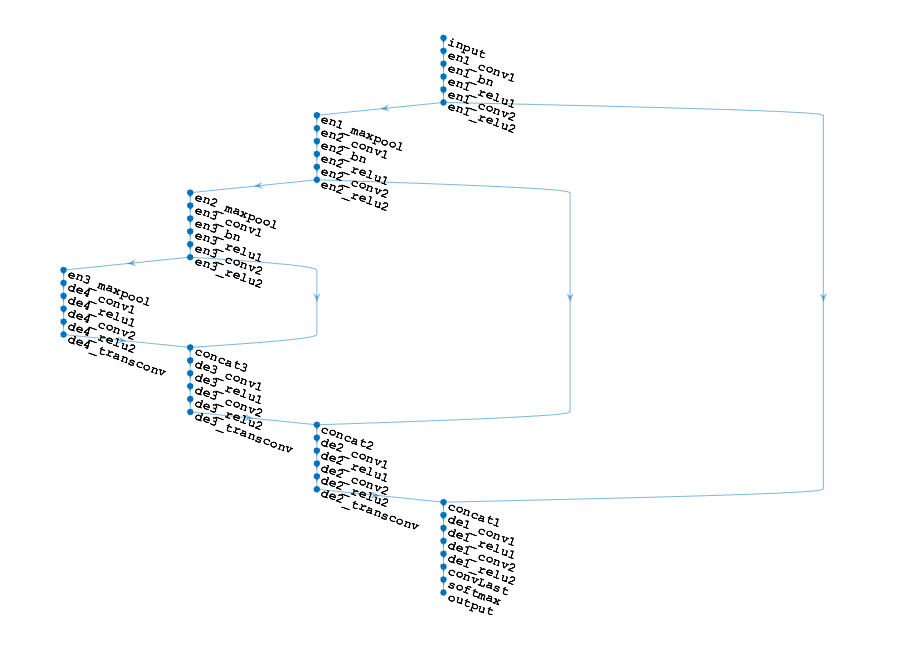
或者,您可以使用createUnet3d辅助函数来创建3 d U-Net层。这个函数是附加到例子作为支持文件。万博1manbetx
lgraph = createUnet3d (inputSize);
绘制层图。
analyzeNetwork (lgraph)
指定培训选项
火车使用“亚当”的网络优化解决。指定hyperparameter设置使用trainingOptions函数。最初的学习速率设置为5,张成的空间逐渐减少的军医培训。
选择= trainingOptions (“亚当”,…“MaxEpochs”,100,…“InitialLearnRate”5的军医,…“LearnRateSchedule”,“分段”,…“LearnRateDropPeriod”5,…“LearnRateDropFactor”,0.95,…“ValidationData”dsVal,…“ValidationFrequency”,400,…“阴谋”,“训练进步”,…“详细”假的,…“MiniBatchSize”,miniBatchSize);
下载Pretrained网络和测试集样本
可选地,下载一个pretrained版本的3 d U-Net和五个样本测试卷和相应的标签从有钱的数据集[3]。pretrained模型和样本数据使您能够执行分割测试数据没有下载完整的数据集或等待网络培训。
trained3DUnet_url =“//www.tianjin-qmedu.com/万博1manbetxsupportfiles/vision/data/brainTumor3DUNet.mat”;sampleData_url =“//www.tianjin-qmedu.com/万博1manbetxsupportfiles/vision/data/sampleBraTSTestSet.tar.gz”;imageDir = fullfile (tempdir,“小鬼”);如果~存在(imageDir“dir”mkdir (imageDir);结束downloadTrained3DUnetSampleData (trained3DUnet_url sampleData_url imageDir);
培训网络
配置选项和数据源的训练后,火车3 d U-Net网络使用trainNetwork函数。训练网络,设置doTraining参数在下面的代码真正的。具有计算功能的CUDA-capable NVIDIA GPU™3.0或更高版本强烈建议进行训练。
如果你保持doTraining参数在下面的代码假,然后返回一个例子pretrained 3 d U-Net网络。
注:培训大约需要60小时在NVIDIA™泰坦X和可以更长时间取决于你的GPU硬件。
doTraining = false;如果doTraining modelDateTime = datestr(现在,“dd-mmm-yyyy-HH-MM-SS”);(网络,信息)= trainNetwork (dsTrain、lgraph选项);保存([“trained3DUNet -”modelDateTime的时代,num2str (maxEpochs)“.mat”),“净”);其他的负载(fullfile (imageDir“trained3DUNet”,“brainTumor3DUNet.mat”));结束

您现在可以使用U-Net语义段脑部肿瘤。
执行分割测试数据
选择测试数据的来源包含地面实况卷和标签进行测试。如果你保持useFullTestSet参数在下面的代码假,那么示例使用五卷进行测试。如果你设置useFullTestSet参数真正的,然后55例使用测试图片选自完整的数据集。
useFullTestSet = false;如果useFullTestSet volLocTest = fullfile (preprocessDataLoc,“imagesTest”);lblLocTest = fullfile (preprocessDataLoc,“labelsTest”);其他的volLocTest = fullfile (imageDir,“sampleBraTSTestSet”,“imagesTest”);lblLocTest = fullfile (imageDir,“sampleBraTSTestSet”,“labelsTest”);一会= [“背景”,“肿瘤”];pixelLabelID = [0 1];结束
作物的中央部分图片和标签大小128 -通过- 128 - 128像素点利用辅助函数centerCropMatReader。这个函数是附加到例子作为支持文件。万博1manbetx的voldsTest变量存储地面实况测试图像。的pxdsTest变量存储地面真理标签。
windowSize = (128 128 128);volReader = @ (x) centerCropMatReader (x, windowSize);labelReader = @ (x) centerCropMatReader (x, windowSize);voldsTest = imageDatastore (volLocTest,…“FileExtensions”,“.mat”,“ReadFcn”,volReader);pixelLabelID pxdsTest = pixelLabelDatastore (lblLocTest,一会,…“FileExtensions”,“.mat”,“ReadFcn”,labelReader);
为每个测试图像,将地面实况图像数量和标签添加到单元格数组。使用的训练网络semanticseg体积预测标签为每个测试函数。
在执行分割之后,后处理预测标签,标签nonbrain体素1,相应的背景。使用测试图像来确定体素不属于大脑。你也可以清理预测标签通过移除岛屿和使用填补漏洞medfilt3函数。medfilt3不支持分类数据,所万博1manbetx以把像素标签iduint8在计算之前。然后,把过滤标签分类数据类型,指定原始像素标签id和类名。
id = 1;而hasdata (voldsTest) disp ([处理测试卷的num2str (id)]) groundTruthLabels {id} =阅读(pxdsTest);{id} =阅读卷(voldsTest);tempSeg = semanticseg ({id}卷,净);%得到non-brain地区面具从测试图像。volMask = {id}卷(:,:,:1)= = 0;% non-brain区域预测的标签设置为背景。(1)tempSeg (volMask) =类名;%对预测标签进行中值滤波。tempSeg = medfilt3 (uint8 (tempSeg) 1);%将过滤标签范畴的。tempSeg =分类(tempSeg pixelLabelID,类名);predictedLabels {id} = tempSeg;id = id + 1;结束
处理测试卷1处理测试卷2处理测试卷3处理测试卷4处理测试卷5
比较对网络预测地面实况
选择一个测试图像的语义分割评价的准确性。从4 - d体积数据中提取第一形态和存储这个3 d体积的变量vol3d。
volId = 2;vol3d = {volId}卷(::,:,1);
显示在蒙太奇中心片地面真理和预测沿深度方向标签。
zID =大小(vol3d, 3) / 2;zSliceGT = labeloverlay (vol3d (:,:, zID) groundTruthLabels {volId} (:,:, zID));zSlicePred = labeloverlay (vol3d (:,:, zID) predictedLabels {volId} (:,:, zID));图的标题(”标签地面真理(左)与网络预测(右)的)蒙太奇({zSliceGT; zSlicePred},“大小”(1 - 2),“BorderSize”5)

显示真实的标签卷使用labelvolshow函数。把背景设置可见性完全透明的背景标签(1)0。因为脑内的肿瘤组织,使大脑的一些像素点透明,因此肿瘤是可见的。做一些脑压透明,指定阈值的数量体积范围[0,1]。所有标准化的体积强度低于这个阈值是完全透明。这个例子设置容量阈值小于1,一些大脑像素仍然可见,给肿瘤的空间位置上下文内的大脑。
图h1 = labelvolshow (groundTruthLabels {volId}, vol3d);h1.LabelVisibility (1) = 0;h1。VolumeThreshold = 0.68;
同样的体积,显示预测的标签。
图h2 = labelvolshow (predictedLabels {volId}, vol3d);h2.LabelVisibility (1) = 0;h2。VolumeThreshold = 0.68;
这张照片显示的结果显示片按顺序在整个体积。

量化细分精度
测量使用的分割精度骰子函数。这个函数计算骰子预测之间的相似系数和地面真理分割。
diceResult = 0(长度(voldsTest.Files), 2);为j = 1:长度(卷)diceResult (j:) =骰子(groundTruthLabels {j}, predictedLabels {j});结束
计算平均骰子点数组测试卷。
meanDiceBackground =意味着(diceResult (: 1));disp ([的平均骰子点数的背景的num2str (j),…“测试卷= 'num2str (meanDiceBackground)])
骰子平均得分的背景5测试卷= 0.99341
meanDiceTumor =意味着(diceResult (:, 2));disp ([“肿瘤在骰子平均分数”num2str (j),…“测试卷= 'num2str (meanDiceTumor)])
骰子平均得分的肿瘤5测试卷= 0.85204
图显示了箱线图可视化数据的骰子跨组五个样本测试卷成绩。图中的红线显示骰子值中值类。蓝盒子的上、下界表明第25和第75百分位数,分别。黑色胡须扩展到最极端的数据点不被认为是离群值。

如果你有统计和机器学习的工具箱™,然后您可以使用箱线图函数可视化骰子分数的统计信息在所有您的测试卷。创建一个箱线图,设置createBoxplot参数在下面的代码真正的。
createBoxplot = false;如果箱线图createBoxplot图(diceResult)标题(“测试集骰子准确性”)xticklabels(类名)ylabel (“骰子系数”)结束
总结
这个例子展示了如何创建和火车3 d U-Net网络进行三维大脑肿瘤分割使用的后代数据集。列车网络的步骤包括:
下载和训练数据进行预处理。
创建一个
randomPatchExtractionDatastore为训练数据到网络中。定义层的3 d U-Net网络。
指定培训选项。
列车网络使用
trainNetwork函数。
3 d U-Net训练后网络或加载pretrained 3 d U-Net网络示例执行一组测试数据的语义分割。由视觉比较评估预测细分到地面真理分割和通过测量骰子预测之间的相似系数和地面真理分割。
引用
[1]Cicek O。,A. Abdulkadir, S. S. Lienkamp, T. Brox, and O. Ronneberger. "3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation." In《医学影像计算和计算机辅助介入的国际会议。雅典,希腊,2016年10月,页424 - 432。
[2]Isensee F。,P. Kickingereder, W. Wick, M. Bendszus, and K. H. Maier-Hein. "Brain Tumor Segmentation and Radiomics Survival Prediction: Contribution to the BRATS 2017 Challenge." In学报BrainLes:国际MICCAI Brainlesion车间。加拿大魁北克市,2017年9月,页287 - 297。
[3]“脑癌”。医学分割Decathalon。http://medicaldecathlon.com/
有钱的数据集是由医学十项全能的4.0使用许可证。所有的保证和陈述都否认;有关详细信息,请参阅许可。MathWorks®已经修改数据集联系在一起下载Pretrained网络和测试集样本这个例子。修改后的样本数据集已经被裁剪区域包含主要是大脑和肿瘤和每个通道已经被减去均值和规范化独立除以标准差剪裁的大脑区域。
[4]Sudre, c . H。,W. Li, T. Vercauteren, S. Ourselin, and M. J. Cardoso. "Generalised Dice Overlap as a Deep Learning Loss Function for Highly Unbalanced Segmentations."深度学习在医学图像分析和多通道学习临床决策支持:第三国际研讨会万博1manbetx。加拿大魁北克市,2017年9月,页240 - 248。
另请参阅
imageDatastore|pixelLabelDatastore|randomPatchExtractionDatastore|semanticseg|trainNetwork|trainingOptions|变换

