对准序列对
这个例子展示了如何从GenBank®中提取一些序列,找到开放阅读框架(orf),然后使用全局和局部对齐算法对序列进行对齐。
从MATLAB®工作空间访问NCBI数据
NCBI网站的众多迷人部分之一是基因和疾病部.本节提供了一个全面介绍了医学遗传学。
在这个例子中,你将会看到与家族黑蒙性白痴病.泰-萨克斯是一种常染色体隐性遗传病,由15号染色体上的一个基因(HEXA,编码氨基己糖酶a的亚基)的两个等位基因突变引起。
HEXA的NCBI参考序列有登录号NM_000520.你可以使用getgenbank函数来检索来自NCBI数据存储库中的序列信息,并将其加载到MATLAB®。
humanHEXA = getgenbank('NM_000520');
通过BLAST搜索或在老鼠基因组中搜索,你可以找到一个正交基因,AK080777是小鼠己糖胺酶a基因的登录号。
mouseHEXA = getgenbank('AK080777');
为了方便起见,以前下载的序列包含在mat -文件中。请注意,公共存储库中的数据经常被管理和更新;因此,当您使用最新的数据集时,此示例的结果可能略有不同。
加载(“hexosaminidase.mat”那“humanHEXA”那“mouseHEXA”)
探索开放阅读框(ORF)
你可以使用这个函数seqshoworfs在人类HEXA基因序列中寻找orf。注意最长的ORF在第一个读帧上。变量中的输出值humanORFs是给出开始和终止密码子的位置为每个阅读框上的所有的ORF的结构。
humanORFs = seqshoworfs (humanHEXA.Sequence)

humanORFs =1×3带有字段的结构数组:开始停止
现在看看在鼠标HEXA基因的ORF。在这种情况下的ORF也是第一框架上。
mouseORFs = seqshoworfs (mouseHEXA.Sequence)

mouseORFs =1×3带有字段的结构数组:开始停止
调整序列
第一步是使用全局序列比对来寻找这些序列之间的相似性。你可以看核苷酸序列之间的比对,但通常更有意义的是看蛋白质序列之间的比对,在这个例子中,我们知道序列是编码序列。使用nt2aa功能,将核苷酸序列转换成相应的氨基酸序列。注意,对于两个序列,HEXA基因都出现在第一个帧中,否则应该使用输入参数框架指定一个替代的编码帧。
humanProtein = nt2aa (humanHEXA.Sequence);mouseProtein = nt2aa (mouseHEXA.Sequence);
在序列之间寻找相似性的最简单方法之一是用点图。
seqdotplot (mouseProtein humanProtein)
警告:匹配矩阵具有比可用的屏幕像素更多的积分。通过在Y的1在X轴和2个因子缩放图像
包含(人类己糖胺酶的); ylabel (“老鼠己糖胺酶”);

使用默认设置,点图有点难以解释,所以您可以尝试稍微严格一点的点图。
seqdotplot (mouseProtein humanProtein 4, 3)
警告:匹配矩阵具有比可用的屏幕像素更多的积分。通过在Y的1在X轴和2个因子缩放图像
包含(人类己糖胺酶的); ylabel (“老鼠己糖胺酶”);
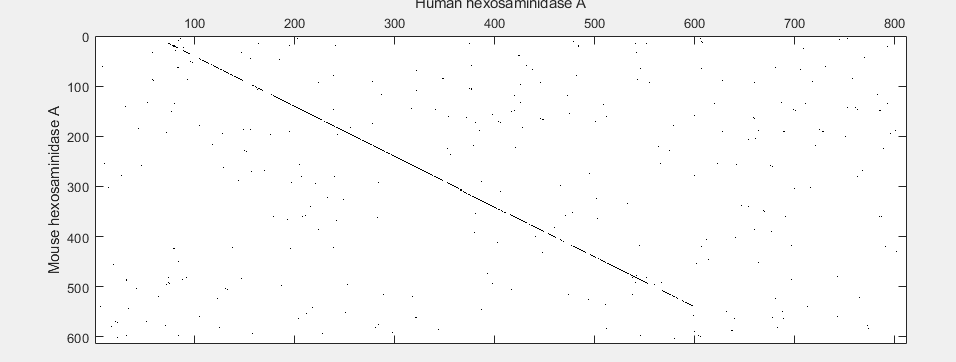
对角线表示可能有一个良好的对齐,因此您现在可以使用该函数查看全局对齐nwalign使用了Needleman-Wunsch算法。
[score, globalAlignment] = nwalign(humanProtein,mouseProtein)(人蛋白,鼠蛋白)
得分= 634.3333
globalAlignment =3 x812 char数组'SCRRPAQSAARSRSLRSRPEVKGQGVGPPGVAGAEPPLVT * FADKSRGRRSPDQGLTWPAPSERGDQRAMTSSRLWFSLLLAAAFAGRATALWPWPQNFQTSDQRYVLYPNNFQFQYDVSSAAQPGCSVLDEAFQRYRDLLFGSGSWPRPYLTGKRHTLEKNVLVVSVVTPGCNQLPTLESVENYTLTINDDQCLLLSETVWGALRGLETFSQLVWKSAEGTFFINKTEIEDFPRFPHRGLLLDTSRHYLPLSSILDTLDVMAYNKLNVFHWHLVDDPSFPYESFTFPELMRKGSYNPVTHIYTAQDVKEVIEYARLRGIRVLAEFDTPGHTLSWGPGIPGLLTPCYSGSEPSGTFGPVNPSLNNTYEFMSTFFLEVSSVFPDFYLHLGGDEVDFTCWKSNPEIQDFMRKKGFGEDFKQLESFYIQTLLDIVSSYGKGYVVWQEVFDNKVKIQPDTIIQVWREDIPVNYMKELELVTKAGFRALLSAPWYLNRISYGPDWKDFYIVEPLAFEGTPEQKALVIGGEACMWGEYVDNTNLVPRLWPRAGAVAERLWSNKLTSDLTFAYERLSHFRCELLRRGVQAQPLNVGFCEQEFEQT * APGTEEGAGCR * MVVEPGFHCILARGRSPLPSCPLPACPCAWRERGRCWRSHSIKSNVAFFYNKHGLPVFKKKSVNGVRVRAQPGWSQCLPLRSFKLRAGNETYSLCAVLPCL * AMSLPSHS * PYSRHLP * SSACSLHFCIISPRRWYMEKDVGAWRCSGQWGGLQTQPGHRRASPPCILIHLPPLELFSFGFLAASILYNHYLNIIKHILFS''|||:||||||| :: ||||||||||:||||||||||:|| :||:||||||||:| |||||| || ||||||:|||:||||||||||| :::|::|| ||:||||||| ||::|:|||||||||||||||||| |||||||||||||||||||||||||||||||:|:|||||||||:|||||||||||||||||||||||||:|||||||||| |||||||||||| ||||:|||||||||||||||||||||||||||||||||||||||||| |||||||||||: ||||||||||||:||:||||:|||:|||||||||||||||||||||||||:|| ||:|||| ||||||||||||||||||:| |||||||||||||||::||||||||||::||:|| |:: :|:|||||||||||||||::|||||||:| ||||||:||||||||||||||||||||||:||||||||||||||||||||::||::: ||::|||||||||:|||:||||::|| |||||||||| | :| : :|| | | || |: :: | | :: | : :| : | :| : : | ::: | | |::| : | | | :| ||::|| | |: | | | :: |: | ' '--------AA------------GR--------G---------A----G-R-------W----------AMAGCRLWVSLLLAAALACLATALWPWPQYIQTYHRRYTLYPNNFQFRYHVSSAAQAGCVVLDEAFRRYRNLLFGSGSWPRPSFSNKQQTLGKNILVVSVVTAECNEFPNLESVENYTLTINDDQCLLASETVWGALRGLETFSQLVWKSAEGTFFINKTKIKDFPRFPHRGVLLDTSRHYLPLSSILDTLDVMAYNKFNVFHWHLVDDSSFPYESFTFPELTRKGSFNPVTHIYTAQDVKEVIEYARLRGIRVLAEFDTPGHTLSWGPGAPGLLTPCYSGSHLSGTFGPVNPSLNSTYDFMSTLFLEISSVFPDFYLHLGGDEVDFTCWKSNPNIQAFMKKKGF-TDFKQLESFYIQTLLDIVSDYDKGYVVWQEVFDNKVKVRPDTIIQVWREEMPVEYMLEMQDITRAGFRALLSAPWYLNRVKYGPDWKDMYKVEPLAFHGTPEQKALVIGGEACMWGEYVDSTNLVPRLWPRAGAVAERLWSSNLTTNIDFAFKRLSHFRCELVRRGIQAQPISVGCCEQEFEQT*A--T--SA--E----HPG-------G------C----CP---------L-SQ-LR--*A--------P---RR-V--LALR-E----Q-VP--G-Q---G-*SFT---------A-SRPGES---T---P----CP---C--APVT--TEKEAGA----GT--GV--Q---*R-----------------------S-MW-HF-------L--'
精炼后半全局比对
除了末端片段外,排列非常好。例如,注意稀疏匹配对在第一个位置。发生这种情况的原因是全局对齐试图强制匹配到所有的端点,并且在某个点上,打开新间隙的惩罚与匹配留数的得分相当。在某些情况下,需要删除在全局对齐的末端添加的间隙惩罚;这允许您更好地匹配这对序列。这种技术通常被称为“半全局”对齐或“glocal”对齐。
[score, globalAlignment] = nwalign(humanProtein,mouseProtein,“全球本土化”,真的)
分数= 1.0413 e + 03
globalAlignment =3 x825 char数组“SCRRPAQSAARSRSLRSRPEVKGQGVGPPGVAGAEPPLVT * FADKSRGRRSPDQGLTWPAPSERGDQR-AMTSSRLWFSLLLAAAFAGRATALWPWPQNFQTSDQRYVLYPNNFQFQYDVSSAAQPGCSVLDEAFQRYRDLLFGSGSWPRPYLTGKRHTLEKNVLVVSVVTPGCNQLPTLESVENYTLTINDDQCLLLSETVWGALRGLETFSQLVWKSAEGTFFINKTEIEDFPRFPHRGLLLDTSRHYLPLSSILDTLDVMAYNKLNVFHWHLVDDPSFPYESFTFPELMRKGSYNPVTHIYTAQDVKEVIEYARLRGIRVLAEFDTPGHTLSWGPGIPGLLTPCYSGSEPSGTFGPVNPSLNNTYEFMSTFFLEVSSVFPDFYLHLGGDEVDFTCWKSNPEIQDFMRKKGFGEDFKQLESFYIQTLLDIVSSYGKGYVVWQEVFDNKVKIQPDTIIQVWREDIPVNYMKELELVTKAGFRALLSAPWYLNRISYGPDWKDFYIVEPLAFEGTPEQKALVIGGEACMWGEYVDNTNLVPRLWPRAGAVAERLWSNKLTSDLTFAYERLSHFRCELLRRGVQAQPLNVGFCEQEFEQT * APGTEEGAGCR * MV-VEPGFHCILA-R - GR - SPLPSCP-LPA-CPCA-WRERGRCWRSHSIK-SNVAFFYNKHGLPVFKKKSVNGVRVRAQPGWSQclplrsfklragnetyslcavlpcl * amslpshs * pysrhlp * ssacslhfciisprrwymekdvgawrcsgqwgglqtqpghrrasppcilihlpplelfsfflasilynhylniikhilfs ' ': || | ||::||| |||||||:| ||||||||| :|| :||:||||||||:| |||||| || ||||||:|||:||||||||||| :::|::|| ||:||||||| ||::|:|||||||||||||||||| |||||||||||||||||||||||||||||||:|:|||||||||:|||||||||||||||||||||||||:|||||||||| |||||||||||| ||||:|||||||||||||||||||||||||||||||||||||||||| |||||||||||:||||||||||||:||:||||:|||:|||||||||||||||||||||||||:|| ||:|||| ||||||||||||||||||:| |||||||||||||||::||||||||||::||:|| |:: :|:|||||||||||||||::|||||||:| ||||||:||||||||||||||||||||||:||||||||||||||||||||::||::: ||::|||||||||:|||:||||::|| |||||||||| ::|: :|| : :: : : :|| | |: | | | : |||| : : : ::: ::|”'------------------------------------------------------------ AAGRGAGRWAMAGCRLWVSLLLAAALACLATALWPWPQYIQTYHRRYTLYPNNFQFRYHVSSAAQAGCVVLDEAFRRYRNLLFGSGSWPRPSFSNKQQTLGKNILVVSVVTAECNEFPNLESVENYTLTINDDQCLLASETVWGALRGLETFSQLVWKSAEGTFFINKTKIKDFPRFPHRGVLLDTSRHYLPLSSILDTLDVMAYNKFNVFHWHLVDDSSFPYESFTFPELTRKGSFNPVTHIYTAQDVKEVIEYARLRGIRVLAEFDTPGHTLSWGPGAPGLLTPCYSGSHLSGTFGPVNPSLNSTYDFMSTLFLEISSVFPDFYLHLGGDEVDFTCWKSNPNIQAFMKKKGF-TDFKQLESFYIQTLLDIVSDYDKGYVVWQEVFDNKVKVRPDTIIQVWREEMPVEYMLEMQDITRAGFRALLSAPWYLNRVKYGPDWKDMYKVEPLAFHGTPEQKALVIGGEACMWGEYVDSTNLVPRLWPRAGAVAERLWSSNLTTNIDFAFKRLSHFRCELVRRGIQAQPISVGCCEQEFEQT * ATSAEHPGGCCPLSQLR * APRRVLALREQVPGQG * SFTASRPGESTPCPCAPVTTEKEAGAGTGVQ * RSMWHFL -------------------------------------------------------------------------------------------------------------------------------------------------------'
通过提取蛋白质序列来改进排列
另一种优化排列的方法是只使用蛋白质序列。注意,对齐的区域由序列中的起始(m -蛋氨酸)和终止(*)氨基酸分隔。如果序列被缩短,只考虑翻译后的区域,那么似乎可以得到更好的对齐。使用找到命令在每个序列中寻找开始氨基酸的索引:
humanStart = find(humanProtein ==“米”, 1)
Humanstart = 70.
查找mouseProtein ==“米”, 1)
mouseStart = 11
同样,使用找到命令查找翻译开始后发生的第一站的索引。特别注意需要特别注意,因为也有在最开始的一站人类普选序列。
humanStop =找到(humanProtein (humanStart:结束)= =‘*’,1) + humanStart - 1
humanStop = 599
Mousestop =查找(MouseProtein(Mousestart:END)==‘*’,1)+ mouseStart - 1
mouseStop = 539
使用这些指数截断序列。
humanSeq = humanProtein(humanStart:humanStop);humanSeqFormatted = seqdisp(humanSeq)
humanSeqFormatted =9 x70 char数组' 1 MTSSRLWFSL LLAAAFAGRA TALWPWPQNF QTSDQRYVLY PNNFQFQYDV SSAAQPGCSV‘61 LDEAFQRYRD LLFGSGSWPR PYLTGKRHTL EKNVLVVSVV TPGCNQLPTL ESVENYTLTI”“121年NDDQCLLLSE TVWGALRGLE TFSQLVWKSA EGTFFINKTE IEDFPRFPHR GLLLDTSRHY”“181年LPLSSILDTL DVMAYNKLNV FHWHLVDDPS FPYESFTFPE LMRKGSYNPV THIYTAQDVK”“241年EVIEYARLRG IRVLAEFDTP GHTLSWGPGI PGLLTPCYSG SEPSGTFGPVNpslnntyef ' '301 mstfflevss vfpdfylhlg gdevdftcwk snpeiqdfmr kkgfgedfkq lesfyiqtll ' '361 divssyggy vvwqevfdnk vkiqpdtiiq vwredipvny mkelelvtka gfrallsapw ' '421 ylnrisygpd vkdfyivepl afegtpeqka lviggeacmw geydntnlv prlwpragav ' '481 aerlwsnklt sdltfayerl shfrcellrr gvqqplnvg fceqfeqt * ' '
Mouseseq = MouseProtein(Mousestart:Mousestop);mouseseqformatted = seqdisp(mouseseq)
mouseSeqFormatted =9 x70 char数组'1 magcrlwvsl llaaalacla talwpwpqyi qtyhrrytly pnnfqfryhv ssaaqagcvv ' ' 61 ldeafrrrn LLFGSGSWPR PSFSNKQQTL gknilvvsvv taecnefpnl esvenytltv ' '121 nddclltvgalrgle tfsqlvwksa egtffinktk ikdfprfphr gvlldtsrhy ' '181 lplssildtl dvmaynkfnv FHWHLVDDSS fpyesftfpe LTRKGSFNPV thiytaqdvk ' '241 evieyarlrg irvlaefdtp ghtlswgpga pglltpcysg SHLSGTFGPVNpslnstydf ' '301 mstlfleiss vfpdfylhlg gdevdftcwk snpniqafmk KKGFTDFKQL esfyiqtlld ' '361 ivsdydkgyv vwqevfdnkv kvrpdtiiqv reempveym lemqditrag frallsapwy ' '421 lnrvkygpdw kdmykvepla fhgtpeqkal viggeacmwg eyvdstnlvp rlwpragava ' '481 erlwssnltt nidfafkrls hfrcelvrrg iqaqpisvgc ceqefeqt * ' '
将这两个序列对齐。
[得分,对准] = nwalign(humanSeq,mouseSeq)
得分= 1.0423E + 03
对齐=3 x530 char数组“MTSSRLWFSLLLAAAFAGRATALWPWPQNFQTSDQRYVLYPNNFQFQYDVSSAAQPGCSVLDEAFQRYRDLLFGSGSWPRPYLTGKRHTLEKNVLVVSVVTPGCNQLPTLESVENYTLTINDDQCLLLSETVWGALRGLETFSQLVWKSAEGTFFINKTEIEDFPRFPHRGLLLDTSRHYLPLSSILDTLDVMAYNKLNVFHWHLVDDPSFPYESFTFPELMRKGSYNPVTHIYTAQDVKEVIEYARLRGIRVLAEFDTPGHTLSWGPGIPGLLTPCYSGSEPSGTFGPVNPSLNNTYEFMSTFFLEVSSVFPDFYLHLGGDEVDFTCWKSNPEIQDFMRKKGFGEDFKQLESFYIQTLLDIVSSYGKGYVVWQEVFDNKVKIQPDTIIQVWREDIPVNYMKELELVTKAGFRALLSAPWYLNRISYGPDWKDFYIVEPLAFEGTPEQKALVIGGEACMWGEYVDNTNLVPRLWPRAGAVAERLWSNKLTSDLTFAYERLSHFRCELLRRGVQAQPLNVGFCEQEFEQT * ' ' |::||| |||||||:| ||||||||| :|| :||:||||||||:| |||||| || ||||||:|||:||||||||||| :::|::|| ||:||||||| ||::|:|||||||||||||||||| |||||||||||||||||||||||||||||||:|:|||||||||:|||||||||||||||||||||||||:|||||||||| |||||||||||| ||||:|||||||||||||||||||||||||||||||||||||||||| |||||||||||:||||||||||||:||:||||:|||:|||||||||||||||||||||||||:|| ||:|||| ||||||||||||||||||:| |||||||||||||||::||||||||||::||:|| |:: :|:|||||||||||||||::|||||||:| ||||||:||||||||||||||||||||||:||||||||||||||||||||::||:::||::|||||||||:|||:||||::|| |||||||||'“MAGCRLWVSLLLAAALACLATALWPWPQYIQTYHRRYTLYPNNFQFRYHVSSAAQAGCVVLDEAFRRYRNLLFGSGSWPRPSFSNKQQTLGKNILVVSVVTAECNEFPNLESVENYTLTINDDQCLLASETVWGALRGLETFSQLVWKSAEGTFFINKTKIKDFPRFPHRGVLLDTSRHYLPLSSILDTLDVMAYNKFNVFHWHLVDDSSFPYESFTFPELTRKGSFNPVTHIYTAQDVKEVIEYARLRGIRVLAEFDTPGHTLSWGPGAPGLLTPCYSGSHLSGTFGPVNPSLNSTYDFMSTLFLEISSVFPDFYLHLGGDEVDFTCWKSNPNIQAFMKKKGF-TDFKQLESFYIQTLLDIVSDYDKGYVVWQEVFDNKVKVRPDTIIQVWREEMPVEYMLEMQDITRAGFRALLSAPWYLNRVKYGPDWKDMYKVEPLAFHGTPEQKALVIGGEACMWGEYVDSTNLVPRLWPRAGAVAERLWSSNLTTNIDFAFKRLSHFRCELVRRGIQAQPISVGCCEQEFEQT *’
打开读框信息也可从输出中获得seqshoworfs命令,但索引是基于核苷酸序列的。使用这些指数来修剪原始的核苷酸序列,然后将它们翻译成氨基酸。
humanPORF = nt2aa (humanHEXA.Sequence (humanORFs (1) .Start (1): humanORFs(1)鸡毛蒜皮(1)));mousePORF = nt2aa (mouseHEXA.Sequence (mouseORFs (1) .Start (1): mouseORFs(1)鸡毛蒜皮(1)));[score, ORFAlignment] = nwalign(humanPORF,mousePORF)
得分= 1042
Orfalignment =.3 x529 char数组“MTSSRLWFSLLLAAAFAGRATALWPWPQNFQTSDQRYVLYPNNFQFQYDVSSAAQPGCSVLDEAFQRYRDLLFGSGSWPRPYLTGKRHTLEKNVLVVSVVTPGCNQLPTLESVENYTLTINDDQCLLLSETVWGALRGLETFSQLVWKSAEGTFFINKTEIEDFPRFPHRGLLLDTSRHYLPLSSILDTLDVMAYNKLNVFHWHLVDDPSFPYESFTFPELMRKGSYNPVTHIYTAQDVKEVIEYARLRGIRVLAEFDTPGHTLSWGPGIPGLLTPCYSGSEPSGTFGPVNPSLNNTYEFMSTFFLEVSSVFPDFYLHLGGDEVDFTCWKSNPEIQDFMRKKGFGEDFKQLESFYIQTLLDIVSSYGKGYVVWQEVFDNKVKIQPDTIIQVWREDIPVNYMKELELVTKAGFRALLSAPWYLNRISYGPDWKDFYIVEPLAFEGTPEQKALVIGGEACMWGEYVDNTNLVPRLWPRAGAVAERLWSNKLTSDLTFAYERLSHFRCELLRRGVQAQPLNVGFCEQEFEQT“|::||| |||||||:| ||||||||| :|| :||:||||||||:| |||||| || ||||||:|||:||||||||||| :::|::|| ||:||||||| ||::|:|||||||||||||||||| |||||||||||||||||||||||||||||||:|:|||||||||:|||||||||||||||||||||||||:|||||||||| |||||||||||| ||||:|||||||||||||||||||||||||||||||||||||||||| |||||||||||:||||||||||||:||:||||:|||:|||||||||||||||||||||||||:|| ||:|||| ||||||||||||||||||:| |||||||||||||||::||||||||||::||:|| |:: :|:|||||||||||||||::|||||||:| ||||||:||||||||||||||||||||||:||||||||||||||||||||::||:::||::|||||||||:|||:||||::|| ||||||||'“MAGCRLWVSLLLAAALACLATALWPWPQYIQTYHRRYTLYPNNFQFRYHVSSAAQAGCVVLDEAFRRYRNLLFGSGSWPRPSFSNKQQTLGKNILVVSVVTAECNEFPNLESVENYTLTINDDQCLLASETVWGALRGLETFSQLVWKSAEGTFFINKTKIKDFPRFPHRGVLLDTSRHYLPLSSILDTLDVMAYNKFNVFHWHLVDDSSFPYESFTFPELTRKGSFNPVTHIYTAQDVKEVIEYARLRGIRVLAEFDTPGHTLSWGPGAPGLLTPCYSGSHLSGTFGPVNPSLNSTYDFMSTLFLEISSVFPDFYLHLGGDEVDFTCWKSNPNIQAFMKKKGF-TDFKQLESFYIQTLLDIVSDYDKGYVVWQEVFDNKVKVRPDTIIQVWREEMPVEYMLEMQDITRAGFRALLSAPWYLNRVKYGPDWKDMYKVEPLAFHGTPEQKALVIGGEACMWGEYVDSTNLVPRLWPRAGAVAERLWSSNLTTNIDFAFKRLSHFRCELVRRGIQAQPISVGCCEQEFEQT”
或者,您可以使用GenBank数据结构中的编码区域信息(CDS)来查找基因的编码区域。
IDX = humanHEXA.CDS.indices;humanCodingRegion = humanHEXA.Sequence(IDX(1):IDX(2));IDX = mouseHEXA.CDS.indices;mouseCodingRegion = mouseHEXA.Sequence(IDX(1):IDX(2));
您还可以从这个结构中获得编码区域的翻译。
humanTranslatedRegion = humanHEXA.CDS.translation;mouseTranslatedRegion = mouseHEXA.CDS.translation;
局部对齐
与其截断序列以寻找更好的对齐,还可以使用局部对齐。这个函数swalign使用Smith-Waterman算法执行本地对齐。这表明整个编码区域的非常好的对准,以及在基因的两端的少数残留物中的少数残留物的合理相似性。
[score, localAlignment] = swalign(humanProtein,mouseProtein)
得分= 1057
localAlignment =3 x547 char数组'RGDQR-AMTSSRLWFSLLLAAAFAGRATALWPWPQNFQTSDQRYVLYPNNFQFQYDVSSAAQPGCSVLDEAFQRYRDLLFGSGSWPRPYLTGKRHTLEKNVLVVSVVTPGCNQLPTLESVENYTLTINDDQCLLLSETVWGALRGLETFSQLVWKSAEGTFFINKTEIEDFPRFPHRGLLLDTSRHYLPLSSILDTLDVMAYNKLNVFHWHLVDDPSFPYESFTFPELMRKGSYNPVTHIYTAQDVKEVIEYARLRGIRVLAEFDTPGHTLSWGPGIPGLLTPCYSGSEPSGTFGPVNPSLNNTYEFMSTFFLEVSSVFPDFYLHLGGDEVDFTCWKSNPEIQDFMRKKGFGEDFKQLESFYIQTLLDIVSSYGKGYVVWQEVFDNKVKIQPDTIIQVWREDIPVNYMKELELVTKAGFRALLSAPWYLNRISYGPDWKDFYIVEPLAFEGTPEQKALVIGGEACMWGEYVDNTNLVPRLWPRAGAVAERLWSNKLTSDLTFAYERLSHFRCELLRRGVQAQPLNVGFCEQEFEQT * APGTEEGAGC'“||||| :: ||||||||||:||||||||||:||:||:||||||||:|||||||||||||||:|||:||||||||||| :::|::|| ||:||||||| ||::|:|||||||||||||||||| |||||||||||||||||||||||||||||||:|:|||||||||:|||||||||||||||||||||||||:|||||||||| |||||||||||| ||||:|||||||||||||||||||||||||||||||||||||||||| |||||||||||: ||||||||||||:||:||||:|||:|||||||||||||||||||||||||:|| ||:|||| ||||||||||||||||||:| |||||||||||||||::||||||||||::||:|| |:: :|:|||||||||||||||::|||||||:| ||||||:||||||||||||||||||||||:||||||||||||||||||||::||::: ||::|||||||||:|||:||||::|| |||||||||| ::|: :||' 'RGAGRWAMAGCRLWVSLLLAAALACLATALWPWPQYIQTYHRRYTLYPNNFQFRYHVSSAAQAGCVVLDEAFRRYRNLLFGSGSWPRPSFSNKQQTLGKNILVVSVVTAECNEFPNLESVENYTLTINDDQCLLASETVWGALRGLETFSQLVWKSAEGTFFINKTKIKDFPRFPHRGVLLDTSRHYLPLSSILDTLDVMAYNKFNVFHWHLVDDSSFPYESFTFPELTRKGSFNPVTHIYTAQDVKEVIEYARLRGIRVLAEFDTPGHTLSWGPGAPGLLTPCYSGSHLSGTFGPVNPSLNSTYDFMSTLFLEISSVFPDFYLHLGGDEVDFTCWKSNPNIQAFMKKKGF-TDFKQLESFYIQTLLDIVSDYDKGYVVWQEVFDNKVKVRPDTIIQVWREEMPVEYMLEMQDITRAGFRALLSAPWYLNRVKYGPDWKDMYKVEPLAFHGTPEQKALVIGGEACMWGEYVDSTNLVPRLWPRAGAVAERLWSSNLTTNIDFAFKRLSHFRCELVRRGIQAQPISVGCCEQEFEQT*ATSAEHPGGC'
互补DNA序列的比对
所有在MATLAB提供的序列比对功能,可定制。例如,通过修改评分的行和列的矩阵可以通过补体,而不是由同一性比对序列。在这种情况下,你可以重新排序NUC44得分矩阵;对补语给予正面分数,否则给予负面分数。小鼠HEXA基因的前30个核苷酸将与它的补体对齐。
[M, info] = nuc44;地图= nt2int (seqcomplement (info.Order))
MAP =1x15 uint8行向量4 3 2 1 6 5 8 7 9 10 14 13 12 11 15
MC = M(:,地图)
MC =15×15-4 -4 -4 -4 5 1 1 -4 -4 1 -1 -1 -1 -4 -2 -4 -4 -4 5 1-4 1-4 1-4 -1 -1 -1 -4-2 -4 5 -4 -4 -4 -4 1 1 1 -4 -1 -4 -1 -1 -2 -4 5 -4 -4 1 -4 -4 1-4 1-4 -1 -1-1 -2 -4 1-4 1-4 -1 -2 -2 -2 -2 -1 -3 -1 -3 -1 1 -4 1-4 -1 -4 -2 -2 -2 -2-3 -1 -3 -1 -1 1 1 -4 -4 -2 -2 -4 -1 -2 -2 -3 -3 -1 -1 -1 -4 -4 11 -2 -2 -1-4 -2 -2 -1 -1 -3 -3 -1 -4 11 -4 -2 -2 -2 -2 -1 -4 -1 -3 -3 -1 -1 1 -4 -4 1-2 -2 -2 -2 -4 -1 -3 -1 -1 -3 -1⋮
[得分,compAlignment] = nwalign(mouseHEXA.Sequence(1:30),......seqcomplement(mouseHEXA.Sequence(1:30)),'SCORINGMATRIX'那......Mc,'字母'那“NT”)
得分= 150.
compAlignment =3 x30 char数组“GCTGCTGGAAGGGGAGCTGGCCGGTGGGCC':::::::::::::::::::::::::::::: 'CGACGACCTTCCCCTCGACCGGCCACCCGG'
关闭所有;

