主要内容
模拟酵母异络蛋白循环
此示例显示如何配置模拟设置,将事件添加到模型中以触发基于时间的更改,保存和绘制模拟结果。此示例使用所描述的模型酵母异质型G蛋白质周期的模型为了说明模型模拟。
加载gprotein.sbproj.项目,包括变量M1,一种偶像生物学®模型对象。
sbioloadproject.GPRotein.
将仿真求解器设置为ode15s.并设置停止时间500.通过编辑索尔弗蒂和停止属性Configset对象与之相关M1模型。
csobj = getConfigset(M1);csobj.solvertype ='ode15s';csobj.stoptime = 500;
指定对所有物种的仿真结果进行指定。
csobj.runtimeOptions.statestolog =.'全部';
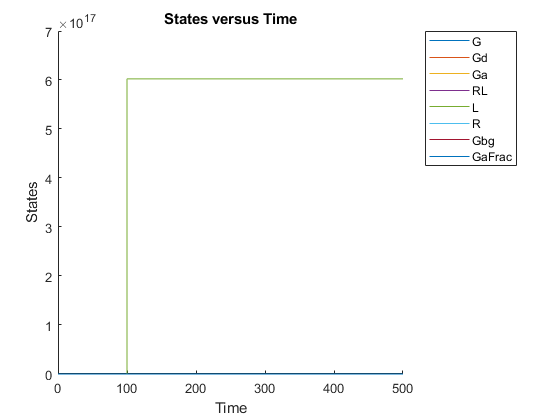
假设配体种类的量L.在仿真开始时是0,但它会增加到特定量= 100.使用SbioSelect.选择命名的物种L.并将其初始金额设置为0.使用addevent.设置所需的事件。
sceniSobj = SbioSelect(M1,'类型'那'物种'那'名称'那'L');sceniSobj.initialAmount = 0;evt = addevent(m1,'时间> = 100'那'l = 6.022e17');
模拟模型。
[t,x,名称] = sbiosmulate(m1);
模拟仿真结果。请注意,当事件在模拟时间100触发事件时,物种L的量增加。由于物种量的宽范围,其他物种的变化不会显示在图中。
绘图(t,x);传奇(名称)Xlabel('时间');ylabel('数量');

要查看其他物种的变化,则没有物种L(第5种)数据的绘图。
图绘图(t,x(:,[1:4 6:8]));图例(名称{[1:4 6:8]});Xlabel('时间');ylabel('数量');

在单独的输出中存储模拟数据的替代方案,例如T.那X, 和名称如上所述,您可以将它们全部存储在一个辛迪塔对象。然后你可以使用SelectByName.提取包含您兴趣的模拟数据的阵列。
Simdata = Sbiosmulate(M1);SBIOPLOT(SIMDATA);
扩张跑1要查看绘制的物种和参数的名称。
simdata_nol = selectbyname(Simdata,{'Ga'那'G'那'GD'那'GAFRAC'那'rl'那'r'}); sbioplot(simdata_noL);


