癌の検出
この例では,タンパク質プロファイルに関する質量分析データを使用して癌を検出するようにニューラルネットワークに学習させる方法を説明します。
はじめに
血清プロテオミクスパターン診断を使用すると,疾患のある患者と疾患のない患者の標本を区別することができます。プロファイルパターンは,SELDI(表面増強レーザー脱着/イオン化)タンパク質質量分析を使用して生成されます。この技術には,悪性腫瘍病理学における臨床診断テストを改善できる可能性があります。
問題:癌の検出
目標は,質量分析デ,タから癌患者と対照患者を区別できる分類器を作成することです。
この例では,分類器による癌患者と対照患者の区別に使用可能な絞り込んだ測定値,つまり”特徴”を選択する方法を使用します。これらの特徴量は,特定の質量/充填量値での选区オン強度レベルです。
デ,タの書式設定
この例で使用されるデタは,ファルovarian_dataset.matに含まれています。“fda-nci臨床プロテオミクスプログラムデ,タバンク”から提供されています。このデタセットの詳細にいては,[1]および[2]を参照してください。
利用序贯并行计算的光谱批处理(生物信息学工具箱)の手順に従って,デ,タファ,ルOvarianCancerQAQCdataset.matを作成します。新しいファesc escルには,変数Y、MZ,およびgrpが含まれています。
Yの各列は,患者から得られた測定値を表します。Yには216人の患者を表す216の列があり,そのう121人は卵巣癌患者で95人は正常な患者です。
Yの各行は,MZに示す特定の質量/充填量値での瑙オン強度レベルを表します。MZには15000個の質量/充填量値があり,Yの各行は特定の質量/充填量値での患者の瑙オン強度レベルを表します。
変数grpは,これらのどの標本が癌患者を表し,どの標本が正常な患者を表すかを示すインデックス情報を保持します。
主な特徴のランク付け
このタスクは,特徴の数が観測値の数を大幅に上回っているにもかかわらず,1つの特徴で正しく分類できる典型的な分類問題です。そのため,目標は,複数の特徴の重み付けを行うと同時に,過適合にならない汎化されたマッピングを生成する方法を適切に学習する分類器を見つけることです。
重要な特徴を見つける簡単な方法は,M / Zの各値が独立であると仮定し,2群t検定の計算を行うことです。rankfeaturesは,最も重要なm / z値のesc escンデックスを返します。たとえば,検定統計量の絶対値によってランク付けされた100個の主机ンデックスを返します。
OvarianCancerQAQCdataset.matを読み込んで,rankfeatures(生物信息学工具箱)を使用して特徴をランク付けし,入力xとして上位100の測定値を選択します。
ind = rankfeatures(Y,grp,“标准”,的tt,“NumberOfIndices”, 100);x = Y(ind,:);
2 .のクラスのタ,ゲットtを以下のように定義します。
T = double(strcmp(“癌症”grp));T = [T;1 - t];
スクリプトの前処理手順と上記の例は,前処理および特徴選択の想定される代表的な手順を示すためのものです。異なる手順またはパラメーターを使用すると,異なる結果が得られたり,結果が改善されたりする場合があります。
[x,t] = ovarian_dataset;谁xt
名称大小字节类属性t 2x216 3456 double x 100x216 172800 double
x内の各列は,216人の異なる患者の1人を表します。
xの各行は,各患者の100個の特定の質量/充填量値でのescオン強度レベルを表します。
変数t216年には2行から成る個の値があり,それぞれの値は癌患者を示す(1,0),または正常な患者を示す[0,1]のいずれかになっています。
フィ,ドフォワ,ドニュ,ラルネットワ,クを使用した分類
いくかの重要な特徴を特定したら,この情報を使用して癌の標本と正常な標本を分類できます。
ニューラルネットワークはランダムな初期重みで初期化されるため,ネットワークの学習後に得られる結果は例を実行するたびに多少異なります。このようなランダム性を回避するには,毎回同じ結果を生成するように乱数シ,ドを設定します。ただし,乱数シ,ドの設定はユ,ザ,独自のアプリケ,ションには不要です。
setdemorandstream (672880951)
5つの隠れ層ニューロンがある1つの隠れ層のフィードフォワードニューラルネットワークを作成し,学習を行います。入力標本およびタ,ゲット標本が自動的に学習セット,検証セット,およびテストセットに分割されます。学習セットは,ネットワ,クに教えるために使用されます。検証セットに対してネットワ,クの改善が続いている限り,学習が続行されます。テストセットを使用することで,ネットワ,クの精度を独立して測定できます。
ネットワークはまだ入力データとターゲットデータに一致するように構成されていないため,入力と出力のサイズは0です。ネットワ,クに学習させるときに,この構成が行われます。
Net = patternnet(5);视图(净)
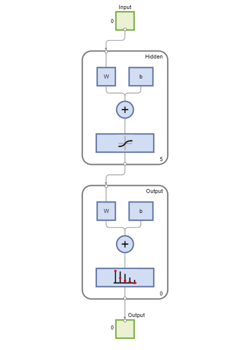
これでネットワ,クの学習の準備が整いました。標本が学習セット,検証セット,およびテストセットに自動的に分割されます。学習セットは,ネットワ,クに教えるために使用されます。検証セットに対してネットワ,クの改善が続いている限り,学習が続行されます。テストセットを使用することで,ネットワ,クの精度を独立して測定できます。
神经网络训练工具を使用すると,学習するネットワークと,学習に使用されているアルゴリズムが表示されます。さらに,学習中には学習の状態が表示され,学習を停止した条件が緑で強調表示されます。
下部にあるボタンを使用すると,便利なプロットを開くことができます。これらのプロットは,学習中および学習後に開くことができます。アルゴリズム名およびプロットボタンの隣のリンクを使用すると,これらに関するドキュメンテーションを開くことができます。
[net,tr] = train(net,x,t);

学習中にネットワーク性能がどのように改善されているかを確認するには,学習ツールの[パフォーマンス]ボタンをクリックするか,関数plotperformを使用します。
性能は,平均二乗誤差で測定され,対数スケ,ルで表示されます。これは,ネットワ,クの学習が進むと急激に低下します。
性能は,学習セット,検証セット,およびテストセットのそれぞれにいて表示されます。
plotperform (tr)

メインデータセットから分割したテスト標本を使用して,学習済みニューラルネットワークをテストできるようになりました。テストデータは学習にまったく使用されていないため,ネットワークをテストするための”標本外“のデータセットとなります。これにより,実際のデ,タでテストした場合にネットワ,クがどの程度の性能かを推定できます。
ネットワ,クの出力は0 ~ 1の範囲にあります。癌患者を示す1または正常な患者を示す0が得られるように出力のしきい値を設定します。
testX = x(:,tr.testInd);testT = t(:,tr.testInd);testY = net(testX);testClasses = testY > 0.5
testClasses =2×32逻辑阵列1 1 1 1 1 1 1 1 1 1 1 1 1 0 0 1 1 0 0 1 1 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 1 0 0 1 1 0 0 1 1 1 1 1 1 1 1 1 0 1
ニューラルネットワークがどの程度データにあてはまるかを測定する方法の1つは,混同プロットです。
混同行列は,正しい分類と正しくない分類の比率を示します。正しい分類は,行列の対角部分の緑の正方形に表示されます。赤い正方形は,正しくない分類を表します。
ネットワ,クが正確な場合,赤い正方形の比率は小さくなり,誤分類がほとんどないことを示します。
ネットワークが正確でない場合は,より長時間の学習を行うか,隠れニューロンを増やしたネットワークの学習を試すことができます。
plotconfusion (testT暴躁的)

正しい分類と正しくない分類の全体的な比率を次に示します。
[c,cm] =困惑(testT,testY);流(正确分类:%f%%\n, 100 * (1 - c));
分类正确率:84.375000%
流('错误分类百分比:%f%%\n', 100 * c);
分类错误百分比:15.625000%
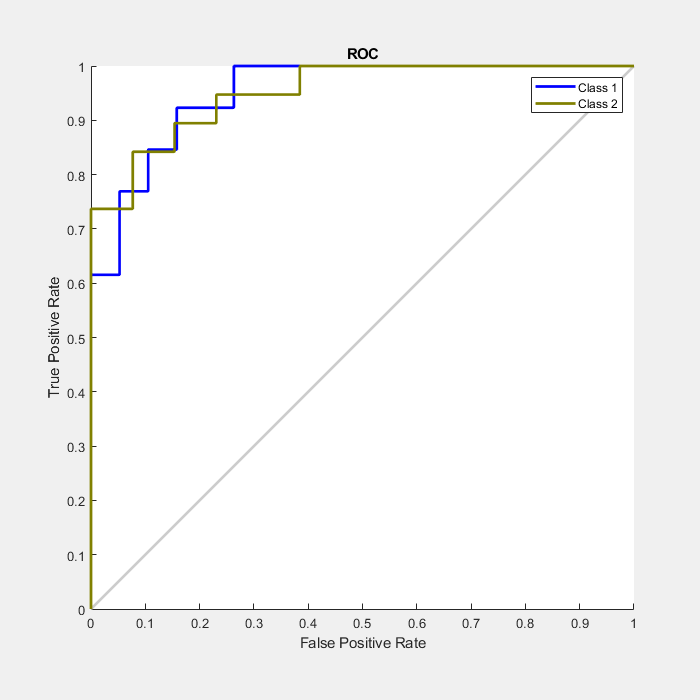
ニューラルネットワークがどの程度データにあてはまるかを測定するもう1つの方法は,受信者動作特性プロットです。このプロットは,出力のしきい値が0 ~ 1の範囲で変化する場合に偽陽性率と真陽性率にどのような関連があるかを示します。
線が左上にあればあるほど,高い真陽性率を得るために受け入れる必要がある偽陽性の数が減少します。最適な分類器とは,線が左下隅から左上隅,右上隅,またはその近くに向かって伸びている分類器です
クラス1は癌患者,クラス2は正常な患者を示します。
plotroc (testT暴躁的)
この例では,ニュ,ラルネットワ,クを癌の検出用分類器として使用する方法を説明しました。分類器の性能を向上させるために,主成分分析などの手法を使用して,ニューラルネットワークの学習に使用されるデータの次元を減らすこともできます。
参考文献
[1] T.P. Conrads等,“高分辨率血清蛋白组学特征用于卵巢检测”,内分泌相关癌症,2004,11,pp. 163-178。
[2] E.F. Petricoin等,“使用血清蛋白质组学模式识别卵巢癌”,柳叶刀,359(9306),2002,pp. 572-577。
