全身基于生理的药代动力学(PBPK)模型
本文档介绍了Simbiology®如本文所述“评估基于生理生理的药代动力学模型用于线形分析”希拉·安妮·彼得斯(Sheila Annie Peters)[1]
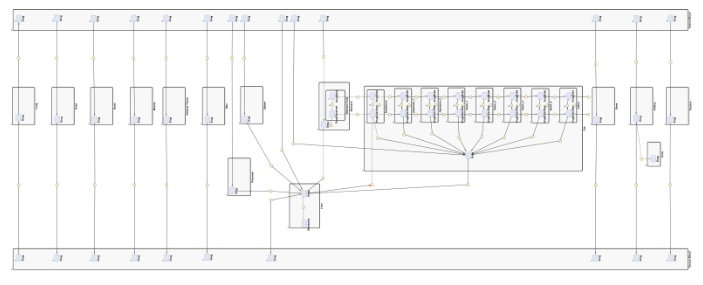
模型描述
此处介绍的通用PBPK模型最初是由Sheila Annie Peters开发的,用于线形分析,通过探索其他吸收或替代性吸收,分布,代谢和排泄(ADME)途径来研究临床PK数据。该模型包括胃肠道吸收的隔室吸收和传输(CAT)模型的扩展版本,包括肠肝回收,肠壁代谢和细胞细胞吸收。组织分区系数是根据Rodgers等人[2] [3]提出的方程计算的。描述阿替洛尔和卡托普利ADME的药物参数作为变体包括在内。使用此Simbiology项目文件包装的其他变体描述了人类禁食,人类美联储和大鼠生理学,并允许易于比例 - 从大鼠PK到人类PK。有关使用此模型的更多信息,请参见“模型”选项卡的描述部分。
参考
[1] Peters,S。A.(2008)。评估基于生理的一般药代动力学模型,用于线形分析。临床药代动力学,47(4),261-75。
[2] Rodgers,T.,Leahy,D。和Rowland,M。(2005)。基于生理的药代动力学建模1:预测中度到突的碱基的组织分布。《药科学杂志》,94(6),1259 - 76。
[3] Rodgers,T。和Rowland,M。(2006)。基于生理的药代动力学建模2:预测酸的组织分布,非常弱的碱基,中性和z连锁。药学杂志,95(6),1238 - 1257
