基于颜色的分割使用K-Means聚类
这个示例展示了如何使用L*a*b* color空间和K-means集群以自动方式分割颜色。
第一步:读取图像
读到hestain.png,这是一张血液毒素和伊红染色的组织图像(H&E)。这种染色方法有助于病理学家区分不同的组织类型。
他= imread (“hestain.png”);imshow(他)、标题('H&E图像');文本(大小(他,2),大小(他,1)+ 15,…“图片由约翰霍普金斯大学艾伦·帕特恩提供”,…“字形大小”7“HorizontalAlignment”,'对');

步骤2:将图像从RGB颜色空间转换为L*a*b*颜色空间
如果忽略亮度的变化,你能看到多少种颜色?有三种颜色:白色、蓝色和粉色。请注意,您可以很容易地从视觉上区分这些颜色。L*a*b*颜色空间(也称为CIELAB或CIEL *a*b*)使您能够量化这些视觉差异。
L*a*b*颜色空间由CIE XYZ三刺激值派生而来。L*a*b*空间由亮度层“L*”、色度层“a*”和色度层“b*”组成,色度层“a*”指示颜色在红绿轴上的位置,色度层“b*”指示颜色在蓝黄轴上的位置。所有的颜色信息都在“a*”和“b*”层中。可以使用欧几里得距离度量来度量两种颜色之间的差异。
转换图像到L*a*b*颜色空间使用RGB2实验室。
实验室he=rgb2lab(he);
步骤3:使用K-Means聚类对“a*b*”空间中的颜色进行分类
集群是一种分离对象组的方法。K-means集群将每个对象视为在空间中有一个位置。它查找分区,使每个集群中的对象尽可能地彼此接近,而其他集群中的对象尽可能地远离。K-means集群要求您指定要分区的集群的数量,并使用距离度量来量化两个对象之间的距离。
由于颜色信息存在于“a*b*”颜色空间中,因此对象是具有“a*”和“b*”值的像素。将数据转换为数据类型单用于imsegkmeans。使用imsegkmeans将对象分为三个簇。
ab=实验室(:,:,2:3);ab=IM2单个(ab);nColors=3;重复群集3次以避免局部极小值nColors pixel_labels = imsegkmeans (ab),“NumAttempts”3);
对于输入的每个对象,imsegkmeans返回与集群对应的索引或标签。用它的像素标签标记图像中的每个像素。
imshow (pixel_labels[])标题(“图像被聚类索引标记”);

步骤4:创建按颜色分割H&E图像的图像
使用pixel_labels,你可以分离对象hestain.png通过颜色,这将产生三个图像。
mask1=像素标签=1;cluster1=he.*uint8(mask1);imshow(cluster1)标题(“群集1中的对象”);

mask2=像素标签=2;cluster2=he.*uint8(mask2);imshow(cluster2)标题(“集群2中的对象”);

mask3=pixel_labels==3;cluster3=he.*uint8(mask3);imshow(cluster3)标题(“集群3中的对象”);

步骤5:分割细胞核
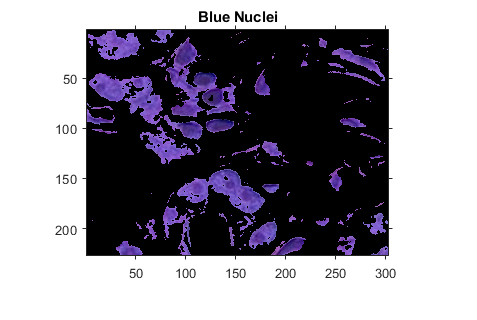
Cluster 3包含蓝色的对象。请注意,有暗蓝色和浅蓝色的对象。你可以使用L*a*b*颜色空间中的“L*”层来区分深蓝色和浅蓝色。细胞核呈深蓝色。
回想一下,“L*”层包含每种颜色的亮度值。使用全局阈值提取集群中像素的亮度值并对其进行阈值imbinarize。面具is_light_blue给出淡蓝色像素的索引。
L = lab_he (:: 1);L_blue = L .* double(mask3);L_blue =重新调节(L_blue);idx_light_blue = imbinarize(非零(L_blue));
复制蓝色物体的蒙版,mask3,然后从遮罩中移除浅蓝色像素。将新遮罩应用到原始图像并显示结果。只有深蓝色的细胞核可见。
blue_idx =找到(mask3);mask_dark_blue = mask3;mask_dark_blue (blue_idx (idx_light_blue)) = 0;蓝色核= he .* uint8(mask_dark_blue);imshow (blue_nuclei)标题(“蓝色细胞核”);