基于顺序并行计算的光谱批量处理
这个例子展示了如何使用单台计算机、多核计算机或计算机集群来预处理大量的质谱分析信号。注意:本示例的最后一部分需要并行计算工具箱™和MATLAB®并行服务器™。
介绍
这个例子展示了在一个或多个目录中包含的一组质谱上建立批处理操作所需的步骤。您可以使用多核计算机或计算机集群顺序地或并行地实现此操作。批处理适用于单程序多数据(SPMD)并行计算模型,最适合于parallel computing Toolbox™和MATLAB®parallel Server™。
预处理信号来自蛋白质表面增强激光解吸/电离飞行时间(SELDI-TOF)质谱。本例中的数据来自FDA-NCI临床蛋白质组学项目数据库。特别地,该示例使用了使用WCX2蛋白阵列生成的高分辨率卵巢癌数据集。有关该数据集的详细描述,请参见[1]和[2]。
设置数据的存储库
此示例假设您已经下载了数据集并将其解压缩到存储库中。理想情况下,应该将数据集放在网络驱动器中。如果工作人员都可以访问网络上相同的驱动器,他们就可以访问驻留在这些共享资源上的所需数据。这是共享数据的首选方法,因为它可以最小化网络流量。
首先,获取数据存储库的名称和完整路径。定义了两个字符串:从本地计算机到存储库的路径,以及集群计算机访问同一目录所需的路径。根据您的网络配置相应地更改两者。
local_repository =“C: / / MassSpecRepository / OvarianCD_PostQAQC /的例子;worker_repository =L: / / MassSpecRepository / OvarianCD_PostQAQC /例子';
对于这个特定的示例,文件存储在两个子目录中:'Normal'和'Cancer'。方法创建包含要处理的文件的列表DIR命令,
cancerFiles = dir([local_repository . dir]“癌症/ * . txt”) normalFiles = dir([local_repository . conf]“正常/ * . txt”])
cancerFiles = 121×1 struct array with fields: name folder date bytes isdir datenum normalFiles = 95×1 struct array with fields: name folder date bytes isdir datenum
把它们放到一个变量中:
文件= [strcat(“癌症/”, {cancerFiles.name})…strcat (“正常/ ', {normalFiles.name}));N = numel(文件)%文件总数
N = 216
顺序批处理
在尝试并行处理所有文件之前,需要使用for循环在本地测试算法。
用需要应用于每个数据集的顺序指令集编写一个函数。输入参数是数据的路径(取决于实际执行工作的机器如何看待它们)和要处理的文件列表。输出参数是预处理信号和M/Z向量。因为预处理后每个谱图的M/Z矢量是相同的,所以只需要存储一次。例如:
类型msbatchprocessing
function [MZ,Y] = msbatchprocessing(repository,files) % % [MZ,Y] = msbatchprocessing(repository,files)的示例函数预处理files files中的%谱图并返回质量/电荷(MZ)和离子%强度(Y)向量。预处理步骤中的硬编码参数已调整为%,以处理示例的高分辨率频谱图。版权所有:The MathWorks, Inc。K = numel(files);Y = 0 (15000,K);%需要预置内存性能的Y的大小MZ = 0 (15000,1);parfor k = 1: k文件=[存储库文件{k}];%读取具有质量电荷和强度值的两列文本文件fid = fopen(file,'r');Ftext = textscan(fid, '%f%f');文件关闭(fid); signal = ftext{1}; intensity = ftext{2}; % resample the signal to 15000 points between 2000 and 11900 mzout = (sqrt(2000)+(0:(15000-1))'*diff(sqrt([2000,11900]))/15000).^2; [mz,YR] = msresample(signal,intensity,mzout); % align the spectrograms to two good reference peaks P = [3883.766 7766.166]; YA = msalign(mz,YR,P,'WIDTH',2); % estimate and adjust the background YB = msbackadj(mz,YA,'STEP',50,'WINDOW',50); % reduce the noise using a nonparametric filter Y(:,k) = mslowess(mz,YB,'SPAN',5); % the mass/charge vector is the same for all spectra after the resample if k==1 MZ(:,k) = mz; end end
注意在函数内部MSBATCHPROCESSING故意使用PARFOR而不是为。批处理通常由迭代之间独立的任务实现。在这种情况下,声明为冷漠可以变成吗PARFOR,创建一系列MATLAB®语句(或程序),可以在顺序计算机,多核计算机或计算机集群上无缝运行,而无需修改。在这种情况下,循环顺序执行,因为您没有创建并行池(假设在并行计算工具箱™偏好中未选中自动创建并行池的复选框,否则MATLAB无论如何都会并行执行)。对于示例目的,只有20个谱图被预处理并存储在Y矩阵。您可以测量MATLAB®完成循环所需的时间抽搐和TOC命令。
Tic repository = local_repository;K = 20;%改为N做所有[MZ,Y] = msbatchprocessing(repository,files(1:K));disp (sprintf (“%d谱图的顺序时间:%f秒”, K, toc))
20个谱图的连续时间:7.725275秒
多核计算机并行批处理
如果您有Parallel Computing Toolbox™,您可以使用本地工作者来并行化循环迭代。例如,如果你的本地机器有四核,你可以使用默认的“本地”集群配置文件启动一个有四个worker的并行池:
POOL = parpool“本地”4);Tic repository = local_repository;K = 20;%改为N做所有[MZ,Y] = msbatchprocessing(repository,files(1:K));disp (sprintf (“与四位当地工作人员平行时间%d谱图:%f秒”, K, toc))
使用“本地”配置文件启动并行池(parpool)…连接并行池(工人数:4)。与4个本地工人并行20个谱图:3.549382秒
停止本地工作线程池:
删除(池)
并行批处理与分布式计算
如果您有Parallel Computing Toolbox™和MATLAB®Parallel Server™,您还可以将循环迭代分发到更多的计算机上。在本例中,集群配置文件'compbio_config_01'链接到6个worker。有关设置和选择并行配置的信息,请参阅并行计算工具箱™文档中的“集群配置文件和计算缩放”。
注意,如果您已经编写了自己的批处理函数,则应该使用cluster profile Manager将其包含在相应的集群配置文件中。这将确保MATLAB®正确地将您的新函数传递给工作人员。您可以使用MATLAB®桌面上的Parallel下拉菜单访问群集配置文件管理器。
POOL = parpool“compbio_config_01”6);Tic repository = worker_repository;K = 20;%改为N做所有[MZ,Y] = msbatchprocessing(repository,files(1:K));disp (sprintf (“与6名远程工作人员进行%d频谱图的并行时间:%f秒”, K, toc))
使用'compbio_config_01'配置文件启动并行池(parpool)…连接并行池(工人数:6)。与6个远程工人并行20个谱图的时间:3.541660秒
停止集群池。
删除(池)
异步并行批处理
上述执行方案都是同步运行的,也就是说,它们会阻塞MATLAB®命令行,直到执行完成。如果希望启动批处理作业并在计算异步运行时访问命令行,则可以手动分发并行任务并稍后收集结果。本例使用与前面相同的集群配置文件。
用一个任务创建一个作业(MSBATCHPROCESSING)。该任务运行在其中一个worker及其内部PARFOR循环分布在并行配置中所有可用的工作线程之间。注意,如果N(谱图数量)远远大于并行配置中可用工作线程的数量,parallel Computing Toolbox™自动平衡工作负载,即使您有一个异构集群也是如此。
抽搐%开始计时Repository = worker_repository;K = n;%做所有的谱图CLUSTER = parcluster()“compbio_config_01”);JOB = createcommunicingjob (CLUSTER,“NumWorkersRange”6 [6]);TASK = createTask(JOB,@msbatchprocessing,2,{repository,files(1:K)});提交(工作)
当作业提交时,您的本地MATLAB®提示符立即返回。一旦并行资源可用,并行作业就会启动。同时,您可以通过检查来监视并行作业任务或工作对象。使用等待方法以编程方式等待任务完成:
等待(任务)的任务。OutputArguments
Ans = 1×2单元格数组{15000×1 double} {15000×216 double}
MZ = task . outpuarguments {1};Y = task . outpuarguments {2};破坏(工作)%完成检索结果disp (sprintf (“与6个远程工作人员进行%d频谱图的并行(异步)时间:%f秒”, K, toc))
6个远程工作者的并行时间(异步)为216个谱图:68.368132秒
后处理
收集完所有数据后,您可以在本地使用它。例如,您可以应用组规范化:
Y = msnorm(MZ,Y,分位数的, 0.5,“限制”(3500 11000),“马克斯”, 50);
创建具有每个谱图类型的分组向量以及索引向量。这种“标记”将有助于对数据集进行进一步分析。
GRP = [repmat({“癌症”},大小(cancerFiles));…repmat ({“正常”},大小(normalFiles));cancerIdx = find(strcmp(grp))“癌症”));元素个数(cancerIdx)"Cancer"子目录下的%文件数
Ans = 121
normalIdx = find(strcmp(grp,“正常”));元素个数(normalIdx)“Normal”子目录下的%文件数
Ans = 95
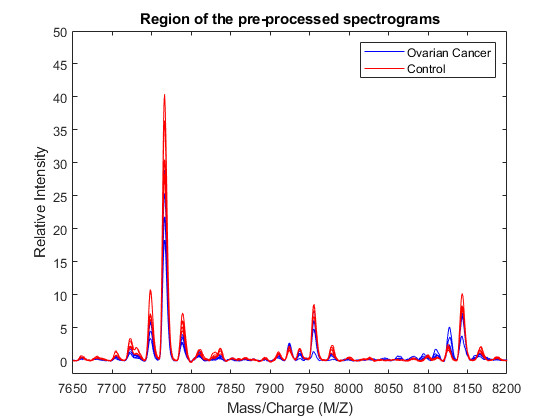
标记数据后,可以使用不同的颜色显示每个类别的一些谱图(在本例中为每组的前五个)。
h = plot(MZ,Y(:,cancerIdx(1:5)),“b”MZ, Y (:, normalIdx (1:5)),“r”);Axis ([7650 8200 -2 50]) xlabel(“质量/电荷(M / Z)”); ylabel (的相对强度)图例(h([1 end]),{卵巢癌的,“控制”})标题(“预处理谱图区域”)
保存预处理的数据集,因为它将在示例中使用识别重要特征和分类蛋白质谱和质谱数据特征的遗传算法搜索。
保存OvarianCancerQAQCdataset.matYMZgrp
免责声明
抽搐-TOC这里以时序为例。顺序和并行执行时间将根据所使用的硬件而有所不同。
参考文献
[1]康拉德,T P, V A Fusaro, S Ross, D Johann, V Rajapakse, B A Hitt, S M Steinberg等,“卵巢癌检测的高分辨率血清蛋白质组学特征”。内分泌相关的癌症,2004年6月,163-78。
[2]petroin, Emanuel F, Ali M Ardekani, Ben A Hitt, Peter J Levine, Vincent A Fusaro, Seth M Steinberg, Gordon B Mills,等。“使用血清中的蛋白质组学模式来识别卵巢癌。”《柳叶刀》359号,没有。9306(2002年二月):572-77。
