探索的核苷酸序列使用序列浏览器应用程序
序列浏览器概述
该序列浏览器集成了许多在生物信息工具箱™工具箱中的序列功能。相反,在MATLAB输入命令的®命令窗口,你可以选择和输入使用的应用程序选项。
导入序列插入序列查看器
分析核苷酸或氨基酸序列时,第一步是将进口序列信息到MATLAB环境。该序列浏览器可以连接到网络数据库,如NCBI和EMBL和读取的信息到MATLAB环境。
下面的过程说明了如何从Web上的NCBI数据库的序列信息。本例使用基因库®登录号NM_000520,这是与戴萨克斯疾病相关的人类基因HEXA。
注意
在公共仓库的数据经常策划和更新;因此,当您使用了最新的序列这个例子的结果可能会略有不同。
在MATLAB命令窗口中输入
seqviewer
或者,单击序列浏览器在应用标签。
该序列浏览器打开没有加载的序列。请注意,窗格右侧和底部是空的。
要检索从NCBI数据库的序列,选择文件>> NCBI下载步骤。
打开从NCBI对话框下载序列。
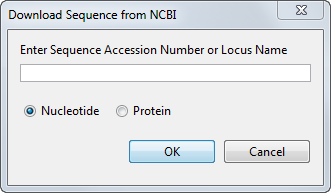
在里面输入序列框,用于NCBI数据库条目类型的登录号,例如,NM_000520。点击核苷酸选项按钮,然后点击好。
MATLAB软件访问NCBI数据库在Web上加载核苷酸对您输入的登录号序列信息,计算出一些基本的统计数据。

查看核苷酸序列信息
在导入的顺序进序列浏览器应用程序,你可以读取存储与序列信息,或者您可以查看的ORF和信用违约掉期的图形表示。
在左窗格中,单击树注释。右侧窗格显示有关序列的一般信息。
现在点击特征。右窗格显示NCBI功能的信息,包括用于基因索引号和任何CDS的序列。
请点击ORF显示在六个阅读框架的开放阅读框的搜索结果。

请点击注释CDS显示的核苷酸序列的蛋白质编码部分。
搜索词
您还可以搜索使用正则表达式特征单词或序列模式。可以进入IUB / IUPAC核苷酸和被自动转换为对应的相应的核苷酸和氨基酸的氨基酸符号。有关符号如何解释的详细信息,请参阅核苷酸转换和氨基酸转化的表seq2regexp。例如,如果你搜索的单词'柏油'与正则表达式选中复选框,应用亮点所有出现'TAA'和'标签'在因为序列R = [AG]。
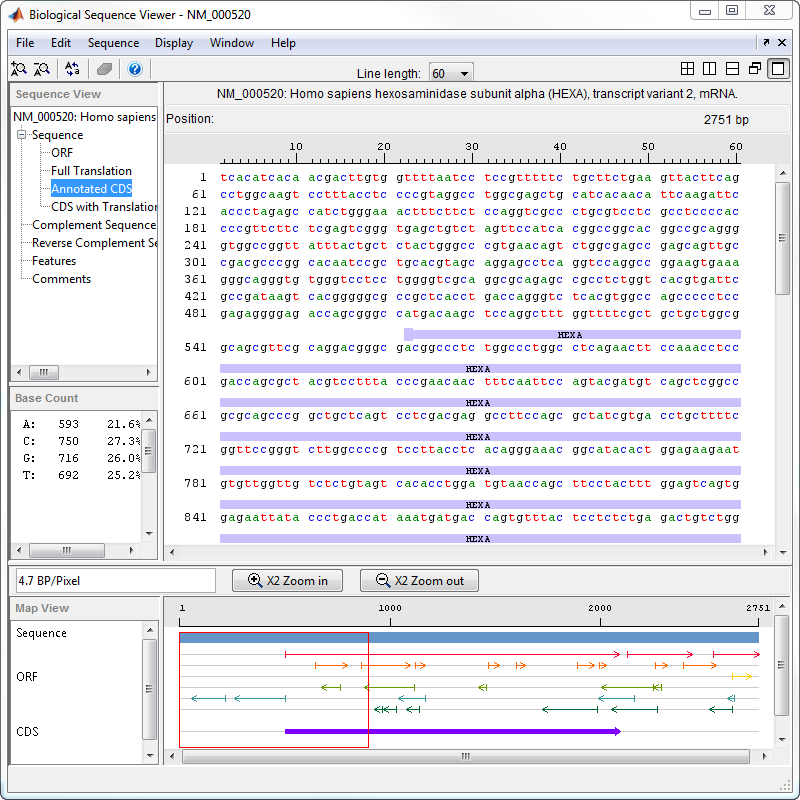
选择序>查找字。
在查找Word对话框,输入一个序列字或图案,例如,ATG,然后单击找。
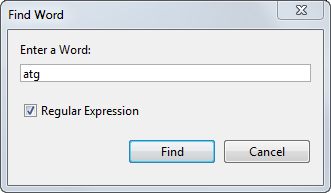
该序列浏览器将搜索并显示所选择的字的位置。
通过单击清除Word中选择按钮清除显示
 在工具栏上。
在工具栏上。
探索开放阅读框
下面的过程示出了如何识别的核苷酸序列的蛋白质编码部分,并将其复制到一个新的图。识别的核苷酸序列编码部分是一种常见的生物信息学的任务。定位序列的编码部分后,可以将其复制到一个新的观点,把它翻译成氨基酸序列,并继续与您的分析。
在左窗格中,单击ORF。
该序列浏览器显示的ORF在右下窗格的六个阅读框架。将光标悬停在一帧至约它的显示信息。

点击阅读框2最长的ORF。
的ORF被突出显示,以指示所选择的序列的一部分。
右键单击选定的ORF,然后选择导出到工作区。在导出到MATLAB工作区对话框中,键入一个变量名,例如,NM_000520_ORF_2,然后单击出口。
该NM_000520_ORF_2变量被添加到工作空间MATLAB。
选择文件>从工作区导入。与导出ORF键入一个变量的名称,例如,NM_000520_ORF_2,然后单击进口。
该序列浏览器在底部的新序列添加一个标签,同时保留原来的顺序打开。

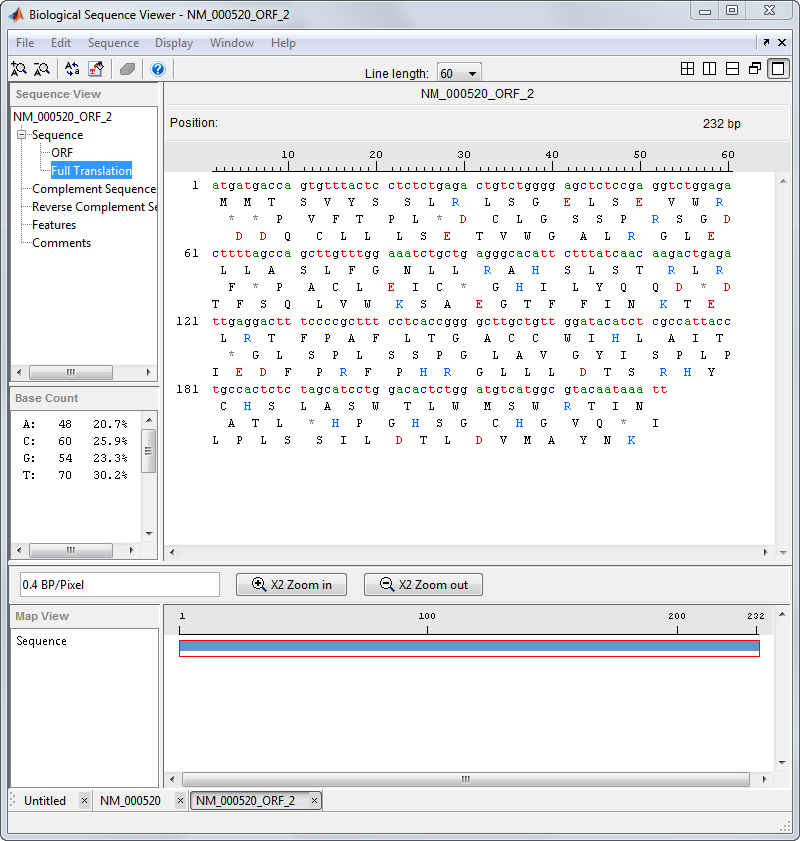
在左窗格中,单击全译。选择显示>氨基酸残基的显示器>单字母代码。
该序列浏览器显示的氨基酸序列的核苷酸序列的下方。
关闭浏览器的序列
关上序列浏览器从MATLAB命令行使用以下语法:
seqviewer( '关闭')












