分类肿瘤图像多分辨率阻塞
这个例子展示了如何将多分辨率整体滑动图片(WSIs)可能不适合在内存中使用一个Inception-v3深层神经网络。
深度学习肿瘤分类方法依赖于数字病理,整个组织幻灯片是成像和数字化。结果WSIs高分辨率,200000 -,- 100000的像素。WSIs通常存储在一个多分辨率格式方便高效的显示,导航和处理的图像。
概述了一个架构的例子使用基于块处理大型WSIs来训练。火车的例子基于Inception-v3网络使用学习技术转移到个人块进行分类正常的或肿瘤。
如果你不想下载训练数据训练网络,然后继续列车网络或下载Pretrained网络这个例子。
准备训练数据
准备培训和验证数据遵循指令预处理多分辨率图像进行训练分类网络。预处理的例子可以节省预处理训练和验证数据存储在一个文件中trainingAndValidationDatastores.mat。
设置的值dataDir变量的位置trainingAndValidationDatastores.mat文件所在的位置。培训和验证数据存储加载到变量dsTrainLabeled和dsValLabeled。
dataDir = fullfile (tempdir,“Camelyon16”);负载(fullfile (dataDir“trainingAndValidationDatastores.mat”))
建立Inception-v3网络层传输的学习
这个示例使用一个Inception-v3网络[2],一个卷积神经网络训练的一百万多幅图像ImageNet数据库[3]。网络是48层深,可以分类图像到1000年对象类别,如键盘,鼠标,铅笔,和许多动物。
的inceptionv3(深度学习工具箱)函数返回一个pretrained Inception-v3网络。Inception-v3需要深度学习工具箱™模型Inception-v3网络支持包。万博1manbetx如果这种支持包没万博1manbetx有安装,那么函数提供一个下载链接。
网= inceptionv3;lgraph = layerGraph(净);
网络的卷积层提取图像特征。最后可学的一层一层和最后的分类输入图像使用图像特征进行分类。这两层包含的信息如何结合成类的概率特性,失去价值,并预测标签。再培训pretrained网络分类的新图像,用新的替换这两个层层适应新的数据集。更多信息,明白了火车深入学习网络对新图像进行分类(深度学习工具箱)。
找到两个图层的名称来代替使用helper函数findLayersToReplace。这个函数是附加到例子作为支持文件。万博1manbetx在Inception-v3,这两层命名为“预测”和“ClassificationLayer_predictions”。
[learnableLayer, classLayer] = findLayersToReplace (lgraph);
这个例子的目的是执行两个类之间的二元分割,肿瘤和正常的。创建一个新的完全连接层两类。取代最后完全连接层与新层。
numClasses = 2;newLearnableLayer = fullyConnectedLayer (numClasses Name =“预测”);lgraph = replaceLayer (lgraph learnableLayer.Name newLearnableLayer);
创建一个新的分类层两类。取代最后分类层与新层。
newClassLayer = classificationLayer (Name =“ClassificationLayer_predictions”);lgraph = replaceLayer (lgraph classLayer.Name newClassLayer);
指定培训选项
使用根均方列车网络传播(RMSProp)优化。指定RMSProp hyperparameter设置通过使用trainingOptions(深度学习工具箱)函数。
减少MaxEpochs一个小数目,因为大量的训练数据使网络达到收敛更快。指定一个MiniBatchSize根据你的GPU内存可用。虽然大mini-batch大小可以使训练速度,更大的规模可以降低网络的泛化能力。集ResetInputNormalization来假为了防止一个完整的阅读训练数据来计算归一化的数据。
选择= trainingOptions (“rmsprop”,…MaxEpochs = 1,…MiniBatchSize = 256,…洗牌=“every-epoch”,…ValidationFrequency = 250,…InitialLearnRate = 1的军医,…SquaredGradientDecayFactor = 0.99,…ResetInputNormalization = false,…情节=“训练进步”);
列车网络或下载Pretrained网络
默认情况下,这个示例下载pretrained版本的训练分类网络使用helper函数downloadTrainedCamelyonNet。pretrained网络可以用来运行整个示例没有等待培训完成。
训练网络,设置doTraining变量在下面的代码真正的。列车网络使用trainNetwork(深度学习工具箱)函数。
火车在一个或多个gpu,如果可用。使用GPU需要并行计算工具箱™和CUDA NVIDIA GPU®®启用。有关更多信息,请参见GPU计算的需求(并行计算工具箱)。
doTraining =假;如果doTraining checkpointsDir = fullfile (dataDir,“检查点”);如果~存在(checkpointsDir“dir”mkdir (checkpointsDir);结束options.CheckpointPath = checkpointsDir;options.ValidationData = dsValLabeled;trainedNet = trainNetwork (dsTrainLabeled、lgraph选项);modelDateTime =字符串(datetime (“现在”格式=“yyyy-MM-dd-HH-mm-ss”));保存(dataDir +“trainedCamelyonNet——”+ modelDateTime +“.mat”,“trainedNet”);其他的trainedCamelyonNet_url =“//www.tianjin-qmedu.com/万博1manbetxsupportfiles/vision/data/trainedCamelyonNet.mat”;dataDir = fullfile (tempdir,“Camelyon16”);downloadTrainedNetwork (trainedCamelyonNet_url dataDir);负载(fullfile (dataDir“trainedCamelyonNet.mat”));结束
下载测试数据
130 WSIs Camelyon16测试数据集。这些图片都有正常和肿瘤组织。每个文件的大小大约是2 GB。
下载测试数据,去Camelyon17网站,点击第一个“CAMELYON16数据集”链接。打开“测试”目录,然后按照以下步骤。
下载“lesion_annotations。邮政”文件。将所有文件解压到指定的目录
testAnnotationDir变量。打开“图像”目录。下载文件到指定的目录
testImageDir变量。
testDir = fullfile (dataDir,“测试”);testImageDir = fullfile (testDir,“图片”);testAnnotationDir = fullfile (testDir,“lesion_annotations”);如果~存在(testDir“dir”mkdir (testDir);mkdir (fullfile (testDir,“图片”));mkdir (fullfile (testDir,“lesion_annotations”));结束
测试数据进行预处理
创建blockedImage对象来管理测试图像
测试图像的文件名。然后,创建一个数组blockedImage对象管理测试图像。每一个blockedImage对象指向相应的图像文件在磁盘上。
testFileSet = matlab.io.datastore.FileSet (testImageDir + filesep +“测试*”);testImages = blockedImage (testFileSet);
设置所有训练数据的空间参考使用setSpatialReferencingForCamelyon16helper函数。这个函数是附加到例子作为支持文件。万博1manbetx的setSpatialReferencingForCamelyon16函数设置WorldStart和WorldEnd属性的blockedImage使用空间对象引用来自TIF文件元数据的信息。
testImages = setSpatialReferencingForCamelyon16 (testImages);
创建组织面具
有效地处理WSI公司数据,创建一个组织面具为每个测试图像。这个过程是一样的一个用于正常训练图像的预处理。有关更多信息,请参见预处理多分辨率图像进行训练分类网络。
normalMaskLevel = 8;testDir = fullfile (dataDir,“测试”);testTissueMaskDir = fullfile (testDir,“test_tissue_mask_level”+ num2str (normalMaskLevel));如果~ isfolder (testTissueMaskDir) testTissueMasks =应用(testImages, @ (bs) im2gray (bs.Data) < 150…BlockSize = (512 512),…= normalMaskLevel水平,…UseParallel = canUseGPU,…DisplayWaitbar = false,…OutputLocation = testTissueMaskDir);保存(fullfile (testTissueMaskDir“testTissueMasks.mat”),“testTissueMasks”)其他的%加载以前保存的数据负载(fullfile (testTissueMaskDir“testTissueMasks.mat”),“testTissueMasks”);结束
组织的面具只有一个级别,小到可以装进记忆。显示组织面具图片浏览器应用程序使用browseBlockedImageshelper函数。这个helper函数附加到的例子作为支持文件。万博1manbetx
browseBlockedImages (testTissueMasks, 1);
地面实况图像预处理肿瘤
指定的分辨率级别肿瘤面具。
tumorMaskLevel = 8;
创建一个肿瘤为每个地面实况面具肿瘤图像使用createMaskForCamelyon16TumorTissuehelper函数。这个helper函数附加到的例子作为支持文件。万博1manbetx每个图像的函数执行这些操作:
阅读(x,y)边界坐标roi带注释的XML文件中。
单独的肿瘤和正常组织的边界坐标roi到单独的细胞阵列。
细胞边界坐标数组转换成一个二进制屏蔽图片使用
polyToBlockedImage函数。在二进制图像ROI表明肿瘤像素和背景像素显示正常组织。像素内肿瘤和正常组织roi都归类为背景。
testTumorMaskDir = fullfile (testDir, (“test_tumor_mask_level”num2str (tumorMaskLevel)]);如果~ isfolder (testTumorMaskDir) testTumorMasks = createMaskForCamelyon16TumorTissue (testImages, testAnnotationDir, testTumorMaskDir tumorMaskLevel);保存(fullfile (testTumorMaskDir“testTumorMasks.mat”),“testTumorMasks”)其他的负载(fullfile (testTumorMaskDir“testTumorMasks.mat”),“testTumorMasks”);结束
概率预测的热图的肿瘤
使用训练分类网络预测的热图为每个测试图像。热图赋予一个概率评分,每一块的肿瘤类。执行这些操作的例子为每个测试图像创建一个热图:
选择块使用
selectBlockLocations函数。包括所有块像素指定至少有一个组织InclusionThreshold名称-值参数为0。批块使用过程
应用函数定义的处理操作predictBlockhelper函数。辅助函数附加到例子作为支持文件。万博1manbetx的predictBlockhelper函数调用预测(深度学习工具箱)概率函数的数据块并返回分数块肿瘤。气管无名动脉瘘管的热图数据写入一个文件使用
写函数。处理后的最终输出所有块的热图显示整个WSI发现肿瘤的概率。
numTest =元素个数(testImages);outputHeatmapsDir = fullfile (testDir,“热图”);networkBlockSize = (299299 3);抽搐为印第安纳州= 1:numTest气管无名动脉瘘管的%检查文件已经存在[~,id] = fileparts (testImages(印第安纳州).Source);输出文件= fullfile (outputHeatmapsDir id +“.tif”);如果~存在(输出文件,“文件”)劳工统计局= selectBlockLocations (testImages(印第安纳州),水平= 1,…BlockSize = networkBlockSize,…掩码= testTissueMasks(印第安纳州),InclusionThreshold = 0);%热图是内存blockedImage对象贝克海姆=应用(testImages(印第安纳州),@ (x) predictBlockForCamelyon16 (x, trainedNet),…水平= 1,BlockLocationSet = bls BatchSize = 128,…PadPartialBlocks = true, DisplayWaitBar = false);气管无名动脉瘘管的%结果写入一个文件写(贝克海姆,输出文件,BlockSize = [512 - 512]);结束结束toc
收集所有的书面的热图的数组blockedImage对象。
heatMapFileSet = matlab.io.datastore.FileSet (outputHeatmapsDir FileExtensions =“.tif”);bheatMapImages = blockedImage (heatMapFileSet);
可视化的热图
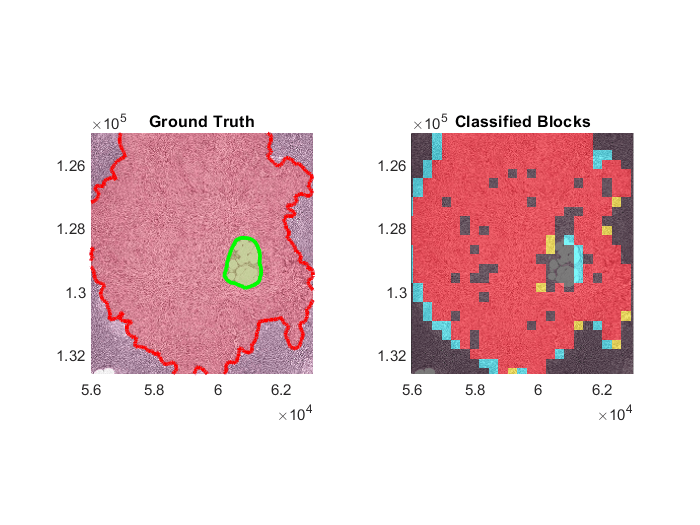
选择一个测试图像显示。左边的图,显示地面真理边界坐标徒手roi使用showCamelyon16TumorAnnotationshelper函数。这个helper函数附加到的例子作为支持文件。万博1manbetx与绿色边界正常区域(如图所示)内可能发生肿瘤区域与一个红色边界(如图所示)。
idx = 27个;图tiledlayout (1、2) nexttile hBim1 = showCamelyon16TumorAnnotations (testImages (idx) testAnnotationDir);标题(“地面实况”)
右边的图,显示测试图像的热图。
nexttile hBim2 = bigimageshow (bheatMapImages (idx)插值=“最近的”);colormap(飞机)
连接轴和放大到一个感兴趣的领域。
linkaxes ([hBim1.Parent hBim2.Parent]) xlim ylim((53982、65269))((122475、133762))标题(“预言的热图”)

测试在特定阈值图像进行分类
对块进行分类,肿瘤或正常的,应用一个阈值的热图概率值。
选择一个阈值概率被归类为上面块肿瘤。理想情况下,你会计算这个阈值使用接受者操作特征曲线(ROC)或precision-recall验证数据集。
打= 0.8;
块在每个测试图像进行分类和计算混淆矩阵使用应用函数定义的处理操作computeBlockConfusionMatrixForCamelyon16helper函数。辅助函数附加到例子作为支持文件。万博1manbetx
的computeBlockConfusionMatrixForCamelyon16helper函数对每个热图:执行这些操作
调整和完善地面真相掩盖的热图的大小相匹配。
应用的热图的阈值。
计算混淆矩阵的所有街区最好的分辨率水平。的混淆矩阵给出了真阳性数(TP),假阳性(FP),真正的负面(TN),假阴性(FN)分类预测。
节省TP的总数量,FP, TN, FN块作为阻塞结构图像。屏蔽的图像返回作为屏蔽图片的数组中的一个元素,
bcmatrix。保存一个数字标记图像的分类预测阻塞的形象。值0,1,2,3对应TN, FP, FN,分别和TP的结果。屏蔽的图像返回作为屏蔽图片的数组中的一个元素,
bcmatrixImage。
为印第安纳州= 1:numTest bcmatrix(印第安纳州),bcmatrixImage{印第安纳}]=应用(bheatMapImages(印第安纳州),…@ (bs tumorMask tissueMask) computeBlockConfusionMatrixForCamelyon16(废话,tumorMask, tissueMask打),…ExtraImages = [testTumorMasks(印第安纳州),testTissueMasks(印第安纳州)]);结束
全球混淆矩阵计算出所有测试图像。
cmArray = arrayfun (@ (c)收集(c), bcmatrix);厘米=[和([cmArray.tp])和([cmArray.fp]);和([cmArray.fn])和([cmArray.tn])];
图表显示混乱的全球混淆矩阵归一化。大多数WSI图像块的正常组织,导致很大一部分真阴性预测。
图confusionchart(厘米,“肿瘤”,“正常”),归一化=“total-normalized”)

可视化分类结果
比较地面实况ROI边界坐标与分类结果。左边的图,显示地面真理边界坐标徒手roi。右边的图,显示测试图像和叠加颜色每一块基于混淆矩阵。真阳性显示为红色,青色的假阳性,假阴性是黄色的,和真正的底片,没有颜色。
假阴性和假阳性出现肿瘤的边缘地区,这表明网络有困难与部分类分类模块。
idx = 27个;图tiledlayout (1、2) nexttile hBim1 = showCamelyon16TumorAnnotations (testImages (idx) testAnnotationDir);标题(“地面实况”)nexttile hBim2 = bigimageshow (testImages (idx));cmColormap = [0 0 0;0 1 1;1 1 0;1 0 0];showlabels (hBim2 bcmatrixImage {idx},…Colormap = cmColormap AlphaData = bcmatrixImage {idx})标题(“分类模块”)linkaxes ([hBim1.Parent hBim2.Parent]) xlim (63000 [56000]) ylim (132600 [125000])
注意:减少肿瘤分类错误的四周,你可以用更少的同质块重新训练网络。当预处理肿瘤块的训练数据集,减少的价值InclusionThreshold名称-值参数。
量化网络预测AUC-ROC曲线
计算通过使用ROC曲线值在不同的阈值computeROCCurvesForCamelyon16helper函数。这个helper函数附加到的例子作为支持文件。万博1manbetx
约打= [1 0.99 0.9::。1 0.05 0];(tpr,玻璃钢,ppv) = computeROCCurvesForCamelyon16 (bheatMapImages、testTumorMasks testTissueMasks,打);
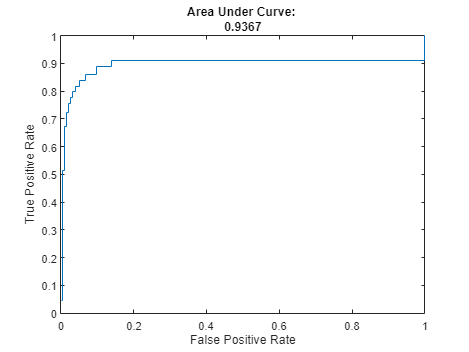
计算曲线下的面积(AUC)度量使用trapz函数。度规返回一个值的范围[0,1],1表示完全模型性能。这个数据集的AUC是接近1。您可以使用AUC来调整培训过程。
图楼梯(玻璃钢、tpr、“-”);ROCAUC = trapz(玻璃钢,tpr);标题([曲线下的面积:“num2str (ROCAUC)]);包含(“假阳性”)ylabel (“真阳性率”)
引用
[1]Ehteshami Bejnordi、Babak Mitko高山病,保罗•约翰内斯·范·迪斯特布拉姆·范·Ginneken尼科Karssemeijer, Geert Litjens,珀斯a·w·m·范德Laak et al。”诊断评估深度学习算法的检测在乳腺癌患者淋巴结转移。”《美国医学会杂志》318年,没有。22岁(2017年12月12日):2199 - 2210。https://doi.org/10.1001/jama.2017.14585。
[2]Szegedy,基督徒,文森特•Vanhoucke谢尔盖•约飞Jonathon Shlens,兹比格涅夫•Wojna。“重新考虑初始架构计算机视觉。“预印本,2015年12月2日提交。https://arxiv.org/abs/1512.00567v3。
[3]ImageNet。https://www.image-net.org。
另请参阅
blockedImageDatastore|blockedImage|blockLocationSet|selectBlockLocations|bigimageshow|trainingOptions(深度学习工具箱)|trainNetwork(深度学习工具箱)
相关的例子
更多关于
- 建立空间引用了图像
- 过程受阻或低分辨率图像有效地使用部分图像万博 尤文图斯
- 处理阻塞图像有效地使用面具
- 创建标记阻止图像roi和面具
- 数据存储深度学习(深度学习工具箱)
- 深度学习层的列表(深度学习工具箱)

