このページは前リリースの情報です。該当の英語のページはこのリリースで削除されています。
深层学习を使用した脳肿疡の3次元セグメンテーション
この例では,3次元U形网ニューラルネットワークに学习させて,3次元医用画像から脳肿疡のセマンティックセグメンテーションを実行する方法を说明します。この例では,3次元U形网ネットワークに学习させる方法を示し,さらに事前学習済みのネットワークも示します。3 次元セマンティック セグメンテーションには、Compute Capability 3.0 以上の CUDA 対応 NVIDIA™ GPU を使用することを強くお勧めします (Parallel Computing Toolbox™ が必要)。
はじめに
セマンティックセグメンテーションでは,イメージの各ピクセルまたは3次元ボリュームのボクセルにクラスでラベル付けします。この例では,深层学习の各种方法を使用して,MRI(磁共振成像)スキャンで脳肿疡のバイナリセマンティックセグメンテーションを実行する方法を说明します。このバイナリセグメンテーションでは,各ピクセルを肿疡または背景としてラベル付けします。
この例では,3次元U形网アーキテクチャ[1]を使用して,脳肿疡のセグメンテーションを実行します.U-Net的は,セマンティックセグメンテーションの分野で一般的になった高速かつ高效率でシンプルなネットワークです。
医用画像のセグメンテーションの课题の1つとして,3次元ボリュームの格纳と处理に必要なメモリの量があります.GPUリソースの制约があるため,全入力ボリュームでネットワークに学习させることは现実的ではありません。この例では,イメージパッチでネットワークに学习させることによって问题を解决します。この例で使用するオーバーラップタイル手法では,テストパッチをつなぎ合わせてセグメント化された完全なテストボリュームにします。この例では,ニューラルネットワークでの畳み込みの有效な部分を使用して,境界アーティファクトを回避します[五]。
医用画像のセグメンテーションのもう1つの课题として,データにクラスの不均衡があり,従来の交差エントロピー损失の使用时に学习の妨げになることがあります。この例では,重み付きマルチクラス骰子损失关数[4]を使用して,问题を解决します。クラスへの重み付けは,骰子スコアに対する大きな领域の影响を无效にするのに役立ち,ネットワークがより小さい领域をセグメント化する方法を学习するのを容易にします。
学习データ,検证データ,テストデータのダウンロード
この例では,小鬼データセット[2]を使用します.BraTSデータセットには,脳肿疡,すなわち最も一般的な原発性悪性脳肿疡である神経胶肿のMRIスキャンが格纳されています。データファイルのサイズは〜7 GBです.BraTSデータセットをダウンロードしない场合,この例の事前学習済みのネットワークとサンプルテストセットのダウンロードの节に进みます。
臭小子データセットを格纳するディレクトリを作成します。
IMAGEDIR =完整文件(TEMPDIR,“小鬼”);如果〜存在(IMAGEDIR,“dir”mkdir (imageDir);结束
臭小子データをダウンロードするには,医疗分割迪卡侬のWebサイトに移動し,[下载数据]リンクをクリックしまTask01_BrainTumourす。”。焦油”ファイル[3]をダウンロードします。変数IMAGEDIRで指定されたディレクトリにTARファイルを解冻します。正常に解冻されると,IMAGEDIRにはimagesTr,imagesTs,およびlabelsTrという3つのサブカテゴリがあるTask01_BrainTumourという名前のディレクトリが含まれます。
750年データセットには個の4次元ボリュームが格納されており,それぞれが3次元イメージのスタックを表します。各4次元ボリュームのサイズは240 x 240 x 155 x 4であり,最初の3つの次元は3次元ボリュームイメージの高さ,幅,奥行に対応します。4番目の次元は異なるスキャンモダリティに対応します。データセットはボクセルラベルを含む484個の学習ボリュームおよび266個のテストボリュームに分割されています。テストボリュームにはラベルがないため,この例ではテストデータを使用しません。代わりに,この例では484個の学習ボリュームを,学習,検証,およびテストに使用される3つの個別のセットに分割します。
学习データと検证データの前处理
より効率的に3次元U-Netネットワークに学習させるには,補助関数preprocessBraTSdatasetを使用してMRIデータを前処理します。この関数は,この例にサポートファイルとして添付されています。
この补助关数は以下の操作を実行します。
主に脳と肿疡を含む领域に合わせてデータをトリミングします。データをトリミングすると,各MRIボリュームの最も重要な部分とそれに対応するラベルを维持しながら,データのサイズが小さくなります。
平均を減算し,トリミングされた脳の領域の標準偏差で除算することにより,各ボリュームの各モダリティを個別に正規化します。
484个の学习ボリュームを400个の学习セット,29个の検证セット,および55个のテストセットに分割します。
データの前处理は,完了するのに约30分かかることがあります。
sourceDataLoc = [IMAGEDIR filesep“Task01_BrainTumour”]。preprocessDataLoc =完整文件(TEMPDIR,“小鬼”,“preprocessedDataset”);preprocessBraTSdataset(preprocessDataLoc,sourceDataLoc);
学習および検証用のランダムパッチ抽出データストアの作成
ランダムパッチ抽出データストアを使用して,ネットワークに学習データを供給し,学習の進行状況を検証します。このデータストアは,グラウンドトゥルースイメージと対応するピクセルラベルデータからランダムパッチを抽出します。パッチは,任意の大きさのボリュームでの学習時にメモリ不足を防ぐための一般的な手法です。
imageDatastoreを作成して3次元イメージデータを格納します.MATファイル形式は非標準イメージ形式であるため,イメージデータを読み取るために垫ファイルリーダーを使用しなければなりません。補助垫ファイルリーダーmatReadを使用できます。この関数は,この例にサポートファイルとして添付されています。
volReader = @(x) matRead(x);volLoc =完整文件(preprocessDataLoc,'imagesTr');volds = imageDatastore(volLoc,…“FileExtensions”,'。垫','ReadFcn',volReader);
ラベルを保存するpixelLabelDatastoreを作成します。
lblLoc = fullfile (preprocessDataLoc,“labelsTr”);类名= [“背景”,“瘤”]。pixelLabelID = [0 1];pxds = pixelLabelDatastore(lblLoc,类名,pixelLabelID,…“FileExtensions”,'。垫','ReadFcn',volReader);
1つのイメージボリュームとラベルをプレビューします。关数labelvolshowを使用して,ラベル付きボリュームを表示します。背景ラベルの可视性(1)を0に設定して,背景を完全に透明にします。
体积=预览(volds);标记=预览(pxds);viewPnl = uipanel(图中,“标题”,“标记训练卷”);HPRED = labelvolshow(标签,体积(:,:,:,1),“父”viewPnl,…'LabelColor',[0 0 0 1 0 0]);hPred.LabelVisibility(1)= 0;

学习イメージとピクセルラベルデータを格纳するrandomPatchExtractionDatastoreを作成します。パッチサイズとして132 X 132 X 132ボクセルを指定します。“PatchesPerImage”を指定して,学习中にボリュームとラベルの各ペアからランダムに配置された16个のパッチを抽出します。ミニバッチサイズとして8を指定します。
patchSize = [132 132 132]。patchPerImage = 16;miniBatchSize = 8;patchds = randomPatchExtractionDatastore(volds,pxds,patchSize,…“PatchesPerImage”,patchPerImage);patchds.MiniBatchSize = miniBatchSize;
同じ手顺に従って,検证イメージとピクセルラベルデータを格纳するrandomPatchExtractionDatastoreを作成します。検证データを使用して,ネットワークが継続的に学习しているか,时间の経过に伴って适合不足や过适合が発生していないかを评価できます。
volLocVal =完整文件(preprocessDataLoc,“imagesVal”);voldsVal = imageDatastore (volLocVal,…“FileExtensions”,'。垫','ReadFcn',volReader);lblLocVal = fullfile (preprocessDataLoc,'labelsVal');pxdsVal = pixelLabelDatastore(lblLocVal,类名,pixelLabelID,…“FileExtensions”,'。垫','ReadFcn',volReader);dsVal = randomPatchExtractionDatastore(voldsVal,pxdsVal,patchSize,…“PatchesPerImage”,patchPerImage);dsVal.MiniBatchSize = miniBatchSize;
关数转变を,补助关数augmentAndCrop3dPatchによって指定されたカスタム前处理演算と共に使用して,学习データと検证データを拡张します。この关数は,この例にサポートファイルとして添付されています。
关数augmentAndCrop3dPatchは以下の操作を実行します。
学习データをランダムに回転および反転させて,学习をさらにロバストにする。この关数では検证データの回転または反転は行われません。
応答パッチをトリミングし,ネットワークの出力サイズを44×44×44ボクセルにする。
数据源=“培训”;dsTrain =变换(patchds,@(patchIn)augmentAndCrop3dPatch(patchIn,数据源));数据源=“验证”;dsVal =变换(dsVal @ (patchIn) augmentAndCrop3dPatch (patchIn数据源));
3次元U-Net層のセットアップ
この例では,3次元U形网ネットワーク[1]を使用します.U-Net的では,最初の一连の畳み込み层に最大プーリング层が点在し,入力イメージの解像度を逐次下げていきます。これらの层に,一连の畳み込み层が続き,その中にアップサンプリング演算处理が点在し,入力イメージの解像度を逐次上げていきます。バッチ正规化层は各RELU层の前に作成されます.U-Net的の名前は,このネットワークが文字「ù」のように対称の形状で描けることに由来しています。
关数unetLayersを使用して,既定の3次元U形网ネットワークを作成します0.2クラスセグメンテーションを指定します。また,有效な畳み込みパディングを指定して,テストボリュームの予测にオーバーラップタイル手法を使用する际に境界アーティファクトを回避します。
inputPatchSize = [132 132 132 4];numClasses = 2;[lgraph, outPatchSize] = unet3dLayers (inputPatchSize numClasses,“ConvolutionPadding”,“有效”);
小さい腫瘍領域をより適切にセグメント化して大きい背景領域の影響を軽減するため,この例ではdicePixelClassificationLayerを使用します。ピクセル分類層を骰子ピクセル分類層に置き換えます。
outputLayer = dicePixelClassificationLayer ('名称',“输出”);lgraph = replaceLayer (lgraph,“分割层”,outputLayer);
この例の学习データと検证データの前处理の節で既にデータが正規化されています。image3dInputLayerでのデータ正規化は不要なため,入力層をデータ正規化が行われない入力層に置き換えます。
inputLayer = image3dInputLayer (inputPatchSize,“归一化”,'没有','名称','ImageInputLayer');lgraph = replaceLayer (lgraph,'ImageInputLayer',inputLayer);
または,深度学习工具箱™のディープネットワークデザイナーアプリを使用して3次元掌中ネットワークを変更できます。
更新された3次元U-Netネットワークのグラフをプロットします。
analyzeNetwork (lgraph)
学习オプションの指定
亚当最适化ソルバーを使用してネットワークに学习させます。关数trainingOptionsを使用してハイパーパラメーター设定を指定します。学习率の初期値は5E-4に设定されており,学习が进むにつれて徐々に减少します.GPUメモリに基づいてMiniBatchSizeプロパティを试すことができます.GPUメモリを最大限に活用するには,バッチサイズを大きくすることより入力パッチを大きくすることを优先します。MiniBatchSizeの値が小さい場合,バッチ正規化層の効果が小さくなることに注意してください。MiniBatchSizeに基づいて初期学習率を微調整します。
选项= trainingOptions(“亚当”,…'MaxEpochs'50,…'InitialLearnRate'5的军医,…“LearnRateSchedule”,“分段”,…“LearnRateDropPeriod”5,…'LearnRateDropFactor',0.95,…'ValidationData',dsVal,…'ValidationFrequency',400,…“情节”,“训练进步”,…“详细”,假,…“MiniBatchSize”,miniBatchSize);
事前学習済みのネットワークとサンプルテストセットのダウンロード
オプションで,事前学习済みバージョンの3次元U形网と5つのサンプルテストボリューム,さらにこれらに対応する臭小子データセットのラベル[3]をダウンロードします。事前学習済みのモデルとサンプルデータを使用すると,データセット全体をダウンロードしたりネットワークが学習するのを待機したりすることなく,テストデータに対してセグメンテーションを実行できます。
trained3DUnet_url =“//www.tianjin-qmedu.com/万博1manbetxsupportfiles/vision/data/brainTumor3DUNetValid.mat”;sampleData_url =“//www.tianjin-qmedu.com/万博1manbetxsupportfiles/vision/data/sampleBraTSTestSetValid.tar.gz”;IMAGEDIR =完整文件(TEMPDIR,“小鬼”);如果〜存在(IMAGEDIR,“dir”mkdir (imageDir);结束downloadTrained3DUnetSampleData(trained3DUnet_url,sampleData_url,IMAGEDIR);
BraTS数据集的预先训练的三维U-Net模型已经存在。样品BraTS测试数据集已经存在。
ネットワークの学習
学習オプションとデータソースを構成した後,関数trainNetworkを使用して3次元U形网ネットワークに学习させます。ネットワークに学习させるには,次のコードで変数doTrainingを真正に设定します。学习には,计算能力3.0以上のCUDA対応英伟达™GPUを使用することを强くお勧めします。
次のコードで変数doTrainingを假のままにしておくと,この例は事前学习済みの3次元U形网ネットワークを返します。
メモ:4つのNVIDIA™泰坦XP的GPUを使用したマルチGPUシステムでの学习には约30时间を要しますご使用のGPUハードウェアによっては,さらに长い时间がかかる可能性もあります。
doTraining = FALSE;如果doTraining modelDateTime = datestr(现在,'DD-MMM-YYYY-HH-MM-SS');[净,信息] = trainNetwork(dsTrain,lgraph,选项);救([“trained3DUNetValid -”modelDateTime'-时代-'num2str(options.MaxEpochs)'。垫'),'净');其他inputPatchSize = [132 132 132 4];outPatchSize = [44 44 44 2];负载(完整文件(IMAGEDIR,'trained3DUNet','brainTumor3DUNetValid.mat'));结束
これで,U-Netを使用して,脳腫瘍を意味ごとにセグメント化できます。
テストデータのセグメンテーションの実行
イメージボリュームのセマンティックセグメンテーションを実行するには,GPUを使用することを強くお勧めします(并行计算工具箱™が必要)。
テスト用のグラウンドトゥルースボリュームおよびラベルを含むテストデータのソースを选択します。次のコードで変数useFullTestSetを假のままにしておくと,この例ではテスト用のボリュームが5つ使用されます。変数useFullTestSetを真正に设定すると,この例ではデータセット全体から选択された55个のテストイメージが使用されます。
useFullTestSet = false;如果使用ltestset volLocTest = fullfile(preprocessDataLoc,'imagesTest');lblLocTest = fullfile (preprocessDataLoc,'labelsTest');其他volLocTest =完整文件(IMAGEDIR,'sampleBraTSTestSetValid','imagesTest');lblLocTest = fullfile (imageDir,'sampleBraTSTestSetValid','labelsTest');类名= [“背景”,“瘤”]。pixelLabelID = [0 1];结束
変数voldsTestは,グラウンドトゥルーステストイメージを格納します。変数pxdsTestは,グラウンドトゥルースラベルを格纳します。
volReader = @(x) matRead(x);voldsTest = imageDatastore (volLocTest,…“FileExtensions”,'。垫','ReadFcn',volReader);pixelLabelID pxdsTest = pixelLabelDatastore (lblLocTest,一会,…“FileExtensions”,'。垫','ReadFcn',volReader);
オーバーラップタイル手法を使用して,各テストボリュームのラベルを予測します。入力サイズがネットワークの出力サイズの倍数になるように各テストボリュームがパディングされて,有効な畳み込みの影響を補正します。オーバーラップタイルアルゴリズムでは,オーバーラップパッチを選択し,関数semanticsegを使用して各パッチのラベルを予測して,パッチを再度組み合わせます。
id = 1;而hasdata (voldsTest) disp ([“加工测试卷”num2str(ID)]);tempGroundTruth =读(pxdsTest);groundTruthLabels {ID} = tempGroundTruth {1};体积{ID} =读(voldsTest);对测试图像使用反射填充。避免不同模式的填充。volSize =尺寸(体积{ID},(1:3));padSizePre =(inputPatchSize(1:3)-outPatchSize(1:3))/ 2;padSizePost =(inputPatchSize(1:3)-outPatchSize(1:3))/ 2 +(outPatchSize(1:3)-mod(volSize,outPatchSize(1:3)));volPaddedPre = padarray(体积{ID},padSizePre,“对称”,“预”);volPadded = padarray (volPaddedPre padSizePost,“对称”,“职位”);[heightPad, widthPad depthPad ~] =大小(volPadded);(高度、宽度、深度、~)=大小(卷{id});tempSeg =分类(0(高度、宽度、深度,'UINT8'),[0; 1],类名);%重叠策略的分割卷。对于k = 1: outPatchSize (3): depthPad-inputPatchSize (3) + 1对于j = 1: outPatchSize (2): widthPad-inputPatchSize (2) + 1对于I = 1:outPatchSize(1):heightPad-inputPatchSize(1)1 =补丁volPadded(I:+ inputPatchSize(1)-1,…Y:J + inputPatchSize(2)-1,…凯西:k + inputPatchSize (3) 1:);patchSeg = semanticseg(补丁,净);tempSeg(我+ outPatchSize (1) 1…j: + outPatchSize (2) 1,…凯西:k + outPatchSize (3) 1) = patchSeg;结束结束结束把多余的软垫部分裁掉。tempSeg = tempSeg(1:高度,1:宽度,1:深度);%除所预测的体积的结果。predictedLabels {ID} = tempSeg;ID = ID + 1;结束
处理测试体积1处理测试体积2处理测试体积3处理测试体积4处理测试体积5
グラウンドトゥルースとネットワークの予测の比较
いずれかのテストイメージを選択して,セマンティックセグメンテーションの精度を評価します。4次元ボリュームデータから最初のモダリティを抽出し,この3次元ボリュームデータを変数vol3dに格納します。
volId = 1;vol3d =体积{volId}(:,:,:,1);
モンタージュに,グラウンドトゥルースラベルおよび予测されたラベルの中心スライスを奥行方向に沿って表示します。
ZID =尺寸(vol3d,3)/ 2;zSliceGT = labeloverlay(vol3d(:,:,ZID),groundTruthLabels {volId}(:,:,ZID));zSlicePred = labeloverlay(vol3d(:,:,ZID),predictedLabels {volId}(:,:,ZID));图蒙太奇({zSliceGT,zSlicePred}“大小”(1 - 2),“BorderSize”5)标题(“标记的地面实况(左)与网络预测模型(右)”)

关数labelvolshowを使用して,グラウンドトゥルースラベル付きボリュームを表示します。背景ラベルの可視性(1)を0に设定して,背景を完全に透明にします。肿疡は脳组织の内部にあるため,肿疡を见られるように脳のボクセルを一部透明にします。脳のボクセルを一部透明にするには,ボリュームのしきい値を [0, 1] の範囲の数値として指定します。このしきい値を下回るすべての正規化ボリューム強度は、完全に透明になります。この例では、脳の内部にある腫瘍の空間的な位置を確認できるように脳のピクセルの一部を表示されたままにするために、ボリュームのしきい値を 1 未満に設定します。
viewPnlTruth = uipanel(图中,“标题”,“地面实况标记卷”);hTruth = labelvolshow (groundTruthLabels {volId}, vol3d,“父”,viewPnlTruth,…'LabelColor',[0 0 0 1 0 0],'VolumeThreshold',0.68);hTruth.LabelVisibility (1) = 0;
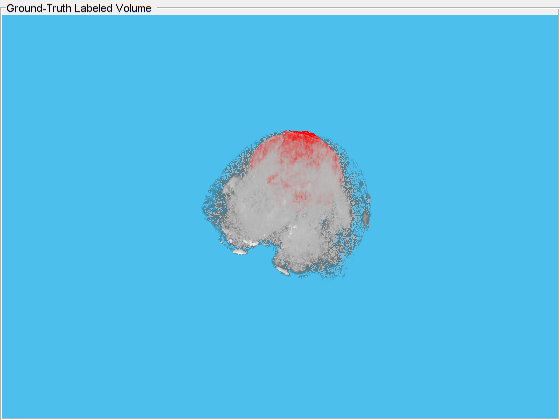
同じボリュームについて,予测ラベルを表示します。
viewPnlPred = uipanel(图中,“标题”,“预测标签卷”);hPred = labelvolshow (predictedLabels {volId}, vol3d,“父”viewPnlPred,…'LabelColor',[0 0 0 1 0 0],'VolumeThreshold',0.68);hPred.LabelVisibility(1)= 0;

このイメージは,一方のボリューム全体のスライスを逐次的に表示した結果を示します。左側はラベル付きのグラウンドトゥルース,右側はネットワーク予測です。

セグメンテーションの精度の定量化
关数骰子を使用してセグメンテーション精度を测定します。この关数は,予测セグメンテーションとグラウンドトゥルースセグメンテーションの骰子类似度系数を计算します。
diceResult = 0(长度(voldsTest.Files), 2);对于j = 1:length(vol) diceResult(j,:) = dice(groundtruthlabel {j}, predictedlabel {j});结束
テストボリュームのセットの平均骰子スコアを计算します。
meanDiceBackground =平均值(diceResult(:,1));DISP([“平均得分骰子背景的跨越”,num2str(J),…'测试卷= 'num2str (meanDiceBackground)])
平均得分骰子背景跨越5个测试体积= 0.9993
meanDiceTumor =意味着(diceResult (:, 2));DISP(["肿瘤的平均骰子点数",num2str(J),…'测试卷= ',num2str(meanDiceTumor)])
平均得分骰子肿瘤跨越5个测试体积= 0.9585
次の図は5つのサンプルテストボリュームのセットの骰子スコアに関する統計量を可視化する箱形图を示します。プロットの赤い線は,クラスの骰子値の中央値を示します。青いボックスの上下の境界はそれぞれ,25日番目の百分位数と75番目の百分位数を示します。黒いひげは,外れ値とは見なされない最も極端なデータ点まで延びます。

统计和机器学习工具箱™がある场合,关数箱形图を使用してすべてのテストボリュームの骰子スコアに关する统计量を可视化できます。箱形图を作成するには,次のコードで変数createBoxplotを真正に設定します。
createBoxplot = FALSE;如果图boxplot(diceResult)“测试仪精度骰子”)xticklabels(类名)ylabel(“骰子系数”)结束
参照
[1]的Çiçek,Ö。,A.阿卜杜勒,S. S. Lienkamp,T.是BrOx和O. Ronneberger。“3D掌中:学习密集体积分割由疏诠释。”在医学影像计算和国际会议论文集计算机辅助干预 - MICCAI 2016。希腊雅典,2016年10月,第424-432页。
[2] Isensee,F.,P. Kickingereder,W.威克,M. Bendszus和K. H.迈尔 - 海因。“脑肿瘤分割和Radiomics生存预测:贡献臭小子2017年的挑战。”在脑组织:国际脑组织损伤研讨会。加拿大魁北克市,2017年9月,287-297页。
[3] “脑肿瘤”。医疗分割Decathalon。http://medicaldecathlon.com/
臭小子データセットは,CC-BY-SA 4.0のライセンスに基づき医疗十项全能によって提供されます。一切の保証および表明を行いません。詳細はライセンスを参照してください.MathWorks®は,この例の事前学習済みのネットワークとサンプルテストセットのダウンロードの节にリンクのあるデータセットに変更を加えています。変更されたサンプルデータセットは主に脳と肿疡を含む领域に合わせてトリミングされており,各チャネルは平均を减算し,トリミングされた脳の领域の标准偏差で除算することにより,个别に正规化されています。
[4] Sudre,C.H.,W.栗,T. Vercauteren,S. Ourselin和M. J.卡多佐。“广义骰子重叠作为极不平衡分割深学习损失函数”。医学图像分析中的深度学习和临床决策支持的多模式学习:第三届国际研讨会万博1manbetx。加拿大魁北克市,2017年9月,240-248页。
[5] Ronneberger, O., P. Fischer,和T. Brox。"U-Net:卷积网络用于生物医学图像分割。"在医学影像计算和国际会议论文集计算机辅助干预 - 2015年MICCAI。德国慕尼黑10月2015年,第234-241。在可用的arXiv:1505.04597。
参考
dicePixelClassificationLayer|imageDatastore|pixelLabelDatastore|randomPatchExtractionDatastore|semanticseg|trainNetwork|trainingOptions|转变

