构建用于评估PK/PD药物疗效的可部署应用程序
作者:Anita Gajjala和Asawari Samant, MathWorks
药代动力学/药效学(PK/PD)和系统药理学模型是评价药物开发的重要工具。使用MATLAB®和SimBiology®在美国,研究人员可以在图形环境中或使用编程工具构建和描述此类模型。他们可以使用临床前数据来数学地描述动态在活的有机体内新候选药物的行为,并根据模型模拟对人类进行预测和推断。然后,他们可以共享这些模型,使临床医生、生物学家和研究人员能够更好地评估各种剂量策略的有效性和安全性指标,并探索不同患者亚群的假设情况。
在某些情况下,直接共享SimBiology模型可能不实际。例如,您的同事可能无法访问或不熟悉MATLAB和SimBiology,或者您可能希望锁定模型及其特性,以便其他用户不会无意中更改它。
MATLAB编译器™允许您将SimBiology模型部署为独立的MATLAB应用程序的一部分。您可以构建一个自定义图形界面,其他研究人员可以使用它来设置和运行模型模拟。然后,您可以编译应用程序,使研究人员无需安装MATLAB或SimBiology即可使用它。
使用由Koch、Walz、Lahu和Schropp开发的肿瘤学模型1作为示例,本文描述了开发和部署应用程序的工作流,该应用程序用于模拟使用SimBiology构建的机制PK/PD模型2.
示例模型和可部署应用程序可用于下载.
肿瘤模型概述
本文中的示例基于Koch等人的肿瘤学模型,该模型用于描述肿瘤在使用单一药物的单一治疗或同时使用两种药物的联合治疗后随时间的生长情况。
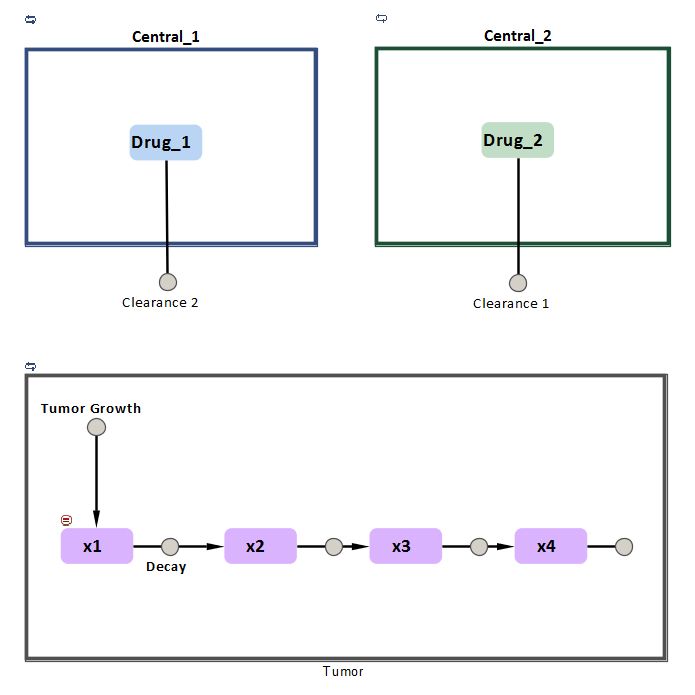
Koch等人肿瘤模型的SimBiology实现如图1所示3..两种药物的血药浓度均采用独立的单室PK模型,并采用线性消除路线。肿瘤生长模型在肿瘤室中表示。衰变反应的速率取决于药物A和药物B的血药浓度和相互作用参数。当药物的血浆浓度降为零时,肿瘤的动态被描述为肿瘤生长过程。肿瘤权重定义为肿瘤生长状态和衰亡状态之和,即x1+ x2+ x3+ x4。
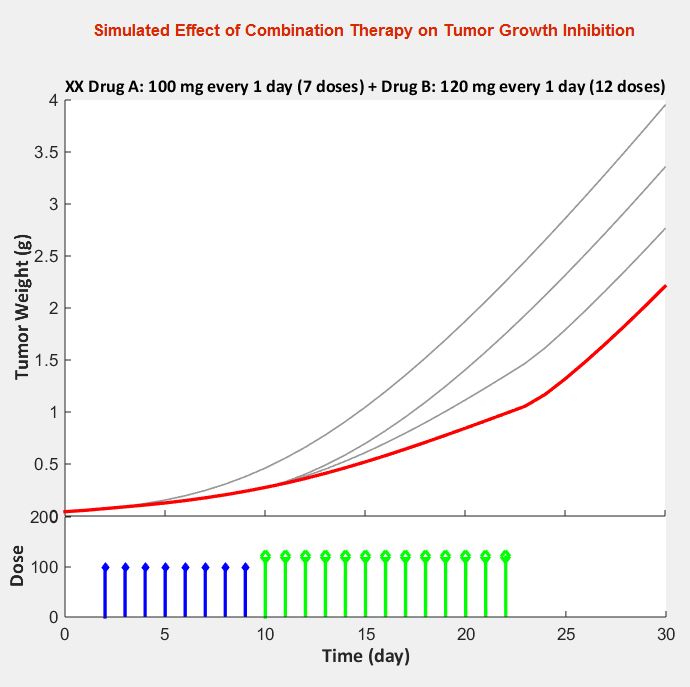
模型模拟使得分析各种联合给药策略对肿瘤抑制的影响成为可能(图2)。
为部署准备模型
为了使用MATLAB编译器共享SimBiology模型,我们首先创建一个导出模型4.
将SimBiology模型加载到MATLAB中sbioloadproject.
> > sbioloadproject (“TumorGrowthModel.sbproj”);加载模型
我们使用出口而且保存来创建导出的SimBiology模型并将其保存为MAT文件,该文件将被加载到部署的应用程序中。
>> exportedModel = export(m1);创建导出模型> >保存modelFile。垫exportedModel保存导出的模型
请注意,导出的SimBiology模型具有一组有限的可访问属性。此外,一些模型特性,包括反应、规则和事件,不能在导出的版本中更改。
构建模型的接口
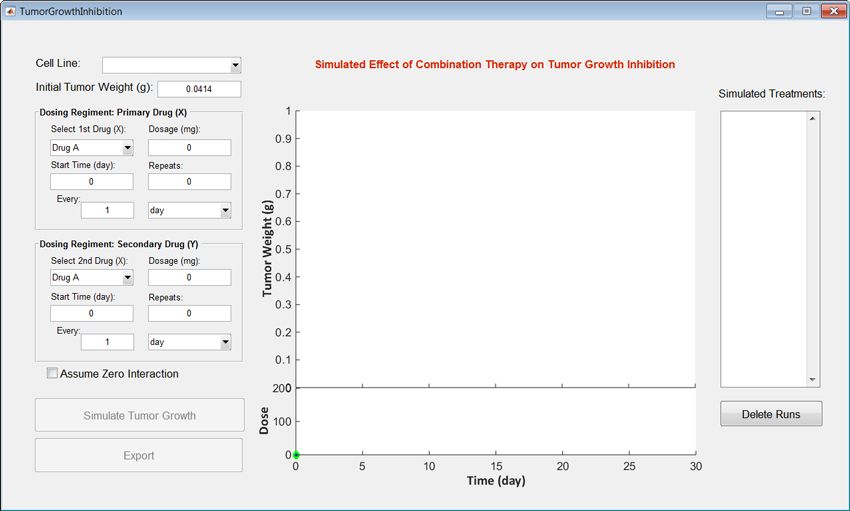
在创建一个导出的SimBiology模型之后,我们构建一个自定义的MATLAB接口。该接口将使无法访问MATLAB的非建模人员和研究人员能够调整模型参数,应用给药计划,并运行模拟(图3)。
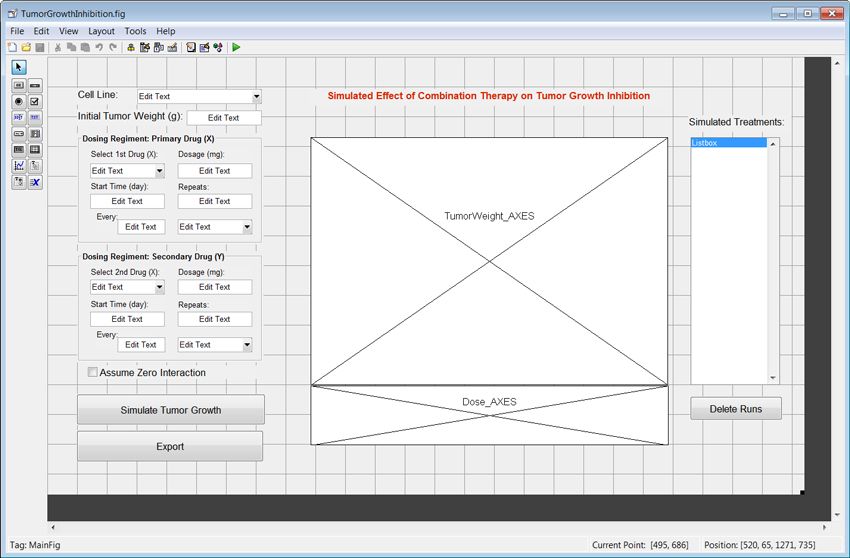
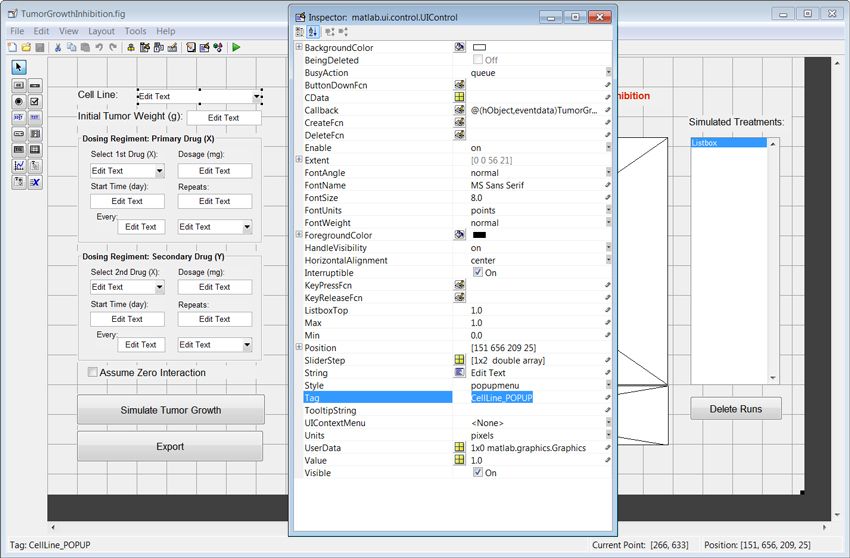
使用MATLAB图形用户界面设计环境(GUIDE)工具,我们通过将组件从面板拖到布局区域来布局界面(图4)。
我们需要修改每个组件的个别属性。例如,我们需要更新标签属性,以便可以轻松地以编程方式访问这些元素。使用命名约定,例如<名称> _ <类型>,提高了代码的可读性(图5)。
编程函数访问导出的SimBiology模型
在基于MATLAB桌面的工作流中,应用程序被编码以访问非导出的SimBiology模型。在为部署做准备时,我们需要更新代码以使用导出的SimBiology模型。
第一步是向MATLAB Compiler指示应用程序使用导出的SimBiology模型。我们通过添加一个指令(通过% #函数在guide生成的MATLAB文件中,TumorGrowthInhibition.m,它链接到我们保存的GUIDE fig -文件。
函数肿瘤生长抑制(varargin)% #函数SimBiology.export.Model
当最终用户与应用程序中的界面控件对象(如菜单或按钮)交互时,特定功能或回调,被调用。接下来,我们将修改回调函数以使用导出的模型。
在示例应用程序中,我们将修改5个回调来为部署模型做准备:TumorGrowthInhibition_OpeningFcn, CellLine_POPUP_Callback,InitialTumorWeight_EDIT_Callback Dosage1_EDIT_Callback,而且SimTumorGrowth_PUSHBUTTON_Callback.
TumorGrowthInhibition_OpeningFcn是示例应用程序中的打开函数。它加载模型并初始化代码中的剂量、模型参数和其他属性。初始版本(未部署)使用sbioloadproject将模型加载到MATLAB中sbiodose或getdose创建或编辑现有的剂量对象。
函数TumorGrowthInhibition_OpeningFcn (hObject eventdata处理,变长度输入宗量)%%创建和初始化剂量——未部署版本%加载项目 Proj = sbioloadproject(“TumorGrowthModel.sbproj”);M1 = project . M1;%服用剂量1D1 = getdose(m1,“药物”);%接受剂量2D2 = getdose(m1,“药物B”);% <剪>
对于部署的应用程序,我们从MAT文件加载导出的模型(而不是使用sbioloadproject).我们使用getdose更新附加到导出模型的现有剂量对象,因为不能将新的剂量对象添加到导出模型。
函数TumorGrowthInhibition_OpeningFcn (hObject eventdata处理,变长度输入宗量)%%创建和初始化剂量-部署版本%加载模型 Proj = load(“modelFile.mat”);m1 = project . exportedmodel;%服用剂量1D1 = getdose(m1,“药物”);%接受剂量2D2 = getdose(m1,“药物B”);% <剪>
CellLine_POPUP_Callback在用户从弹出式菜单中选择单元格行时调用。这个函数负责根据用户的选择适当地设置细胞系参数。在模型的非导出版本中,这是通过选择适当的变量来完成的sbioselect.
函数CellLine_POPUP_Callback (hObject eventdata处理)%%设置适当的细胞系名称不变量——非部署版本 cellLine = sbioselect(模型,“类型”,“变异”,“名字”, cellLineName);% <剪>
SimBiology模型变量不能与导出的模型一起使用。对于导出模型,我们使用ValueInfo而且getIndex获取和设置组成模型变量的单个模型参数。
函数CellLine_POPUP_Callback (hObject eventdata处理)%%设置适当的细胞系参数—已部署版本%获取当前L1和L0值 L1 =模型。ValueInfo(getIndex(model,“L1”));L0 =模型。ValueInfo(getIndex(model,“10”));开关cellLineName情况下“细胞系1号”10。InitialValue = 0.141;L1。InitialValue = 0.282;情况下“细胞系2”10。InitialValue = 0.142;L1。InitialValue = 0.284;情况下“3号细胞系”10。InitialValue = 0.143;L1。InitialValue = 0.286;否则%结束% <剪>
InitialTumorWeight_EDIT_Callback当用户指定初始肿瘤重量(w0).就像CellLine_POPUP_Callback,该回调的非部署版本使用sbioselect在模型中设置参数值。
函数InitialTumorWeight_EDIT_Callback (hObject eventdata处理)%%在模型—非部署版本中设置w0的值 W0 = sbioselect(模型,“类型”,“参数”,“名字”,“w0”);w0。值= str2double(get(hObject,“字符串”));% <剪>
与CellLine_POPUP_Callback,我们使用ValueInfo而且getIndex为部署的应用程序设置导出的SimBiology模型中的值。
函数InitialTumorWeight_EDIT_Callback (hObject eventdata处理)%%在模型部署版本中设置w0的值 W0 =模型。ValueInfo(getIndex(model,“w0”));w0。InitialValue = str2double(get(hObject,“字符串”));% <剪>
Dosage1_EDIT_Callback当用户指定剂量时间单位时调用。此回调函数更新剂量对象。最初,这是通过设置TimeUnits而且时间间隔直接属性。
函数Dosage1_EDIT_Callback (hObject eventdata处理)%%设置剂量对象的设置——非部署版本%直接设置TimeUnits和Interval dose1。TimeUnits = TimeUnits;dose1。Interval = str2double(get(句柄。EveryValue1_EDIT,“字符串”));将开始时间从一天转换为新的时间单位dose1。StartTime = sbiounitcalculator(“天”、dose1.TimeUnits str2double (get(处理。StartTime1_编辑,“字符串”)));% <剪>
在部署的应用程序中,TimeUnits的只读属性是剂量对象,因此不能直接设置。我们使用sbiounitcalculator&将剂量间隔转换为剂量的现有时间单位。
函数Dosage1_EDIT_Callback (hObject eventdata处理)%%设置剂量对象的设置——部署版本%不能设置TimeUnits;使用sbiounitcalculator转换Interval dose1。Interval = sbiounitcalculator(“天”、dose1.TimeUnits str2double (get(处理。EveryValue1_编辑,“字符串”)));将开始时间从一天转换为新的时间单位dose1。StartTime = sbiounitcalculator(“天”、dose1.TimeUnits str2double (get(处理。StartTime1_编辑,“字符串”)));% <剪>
SimTumorGrowth_PUSHBUTTON_Callback当用户单击模拟肿瘤生长按钮。该回调使用适当的参数值和给药计划运行模拟,以计算肿瘤重量随时间的演变。在模型的非部署版本中,它调用sbiosimulate.
函数SimTumorGrowth_PUSHBUTTON_Callback (hObject eventdata处理)%%模拟模型并获得肿瘤剖面-未部署版本 Sd = sbiosimulation(模型,”(cellLine phiVar]、[dose1 dose2]);[currResults.Time, currResults。肿瘤]= selectbyname(sd,' w ');% <剪>
对于导出的模型,我们进行替换sbiosimulate与模拟.
函数SimTumorGrowth_PUSHBUTTON_Callback (hObject eventdata处理)%%模拟模型并获得肿瘤剖面-部署版本 sd =模拟(模型,initValues,[dose1,dose2]);[currResults.Time, currResults。肿瘤]= selectbyname(sd,' w ');% <剪>
设置报表生成
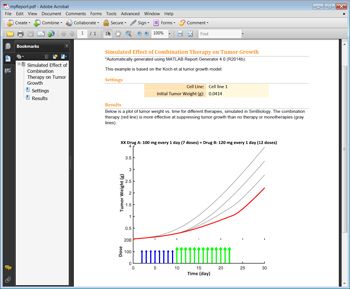
使用MATLAB Report Generator™,我们生成一个文档,捕获模型的PK/PD响应、模型参数和给药计划。我们的应用程序将生成一个名为“myreport.pdf”的PDF文档。此回调使用MATLAB Report Generator 4.0 (R2014b)中提供的新的文档对象模型(DOM) API,该API允许我们以编程方式构建报告。
%——在Export_PUSHBUTTON中按下按钮时执行。函数Export_PUSHBUTTON_Callback(hObject, eventdata, handles)% hObject句柄到Export_PUSHBUTTON(见GCBO) % eventdata保留-在MATLAB的未来版本中定义%句柄结构与用户数据(见GUIDATA) %%生成报告%%%%%%初始化%%%%%% %导入DOM设置 进口mlreportgen.dom。*;使它可部署使用MATLAB编译器makeDOMCompilable ();%创建文档rpt =文档(“myReport”,“pdf”);%%%%%%创建主节%%%%%% %h =标题(2,“联合治疗对肿瘤生长的模拟效果”);b = Border();b.BottomStyle =“单一”;b.BottomColor =浅灰色的;b.BottomWidth =“1分”;h. style = [h.]风格{颜色(“DarkOrange”), b});追加(rpt, h);p =追加(rpt,段落(*使用MATLAB报告生成器4.0 (R2014b)自动生成。));p.Style = {FontFamily(“天线”)、字形大小(“10 pt”)};%%%%%%创建设置节%%%%%% % tableData ={…“细胞系:”, cellLineName {1};初始肿瘤重量(g):initialTumorWeight};table = table (size(tableData,1));追加(rpt、表);%%%%%%创建结果节%%%%%% % hFig =图;肿瘤重量轴hAxes = copyobj(处理。TumorWeight_AXES hFig);集(hax,“单位”,“归一化”);集(hax,“位置”,[0.15 0.30 0.7 0.6]);%剂量轴hAxes = copyobj(处理。Dose_AXES hFig);集(hax,“单位”,“归一化”);集(hax,“位置”,[0.15 0.15 0.7 0.15]);以英寸为单位设置尺寸集(hFig,“单位”,“英寸”);Pos = get(hFig,“位置”);集(hFig,“位置”,[Pos(1:2) 6 5]);集(hFig,“PaperPosition”,[0 0 6 5]);打印并附加到报告打印(hFig“-dmeta”,“plot1.emf”);img =图片(“plot1.emf”);img。宽度=“6”;img。身高=“5”;追加(rpt,段落(img));%%查看报表关闭(rpt);rptview (“myReport”,“pdf”);% <剪>
在MATLAB中测试应用程序
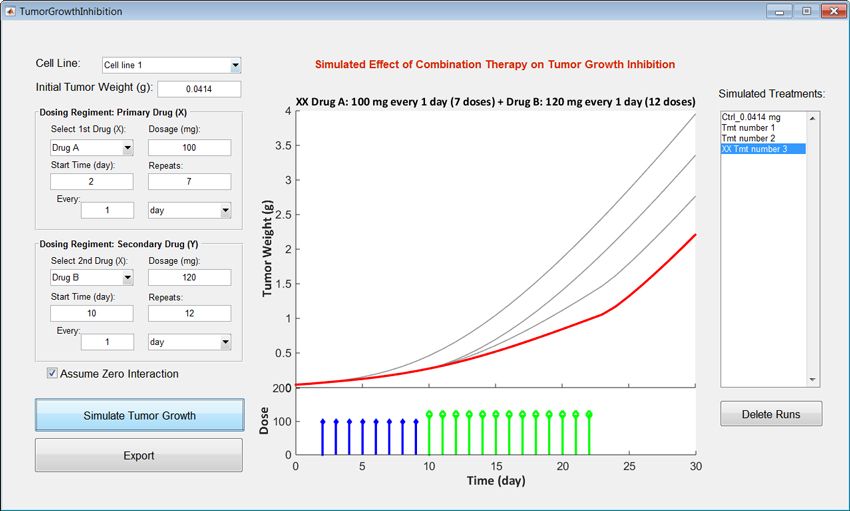
我们执行TumorGrowthInhibition命令在MATLAB中启动应用程序。当应用程序启动时,我们指定细胞系、初始肿瘤重量和给药计划,然后单击模拟肿瘤生长按钮,开始模拟。
该图显示了四种不同处理的模拟结果(图6)。
点击出口按钮,生成显示结果的PDF文件(图7)。
我们现在准备创建一个独立的应用程序,可以分发给没有安装MATLAB的研究人员。
部署应用程序
下面的MATLAB命令定位所有文件和路径依赖项,并调用MATLAB编译器命令来编译TumorGrowthInhibition.m部署申请:
注意,这假设所有其他适用的文件都在path上additionalFiles = [{“modelFile.mat”,“Summary.rpt”}……exportedModel.DependentFiles];为了本例的目的,我们以编程方式构造|mcc| %命令:mccCommand = [“mcc -m TumorGrowthInhibition.m”...sprintf (' -a %s', additionalFiles {:}));%执行|mcc|命令。eval (mccCommand)
一旦编译完成,就会生成一个可执行文件(.exe),用于MATLAB编译运行时(MCR)的分发。MCR是一组共享库,允许用户在没有安装MATLAB的计算机上执行已编译的MATLAB应用程序。
摘要:应用工作流
您可以使用本文中概述的过程,将您自己的SimBiology模型之一部署为独立应用程序。首先在MATLAB中加载模型,并使用出口而且保存命令。接下来,使用GUIDE构建一个简单的接口,并编程回调函数通过接口控制元素访问SimBiology模型。最后,在MATLAB中测试应用程序后,使用MATLAB编译器编译它,并在没有安装MATLAB的计算机上重新测试生成的可执行文件。
1Koch, G., Walz A., Lahu, G.和Schropp, J.(2009)肿瘤生长模型和联合治疗的抗癌效果。药物动力学与药效学杂志36:179-197。
2此工作流中的第一步是构建模型并对其进行描述。这一步是由Koch等人完成的,虽然本文提供了该方法的概述,但没有详细描述。看到在SimBiology中构建模型进一步了解这一步。
3.基本的速率方程没有显示在图1中。请参阅科赫等人或可下载的SimBiology应用程序获取模型和实现的详细信息。
4SimBiology项目文件(*.sbproj)与MATLAB编译器不兼容。
发布于2014 - 92233v00