我怎样才能输出k - means之间的差异和模糊c均值dermoscopy图片吗?
2视图(30天)
显示旧的评论
我曾经在k - means和fuzzy-cmeans功能。代码结构是相同的。当我运行代码几个图片我得到了相同的结果的方法。我也有错误使用模糊c重塑,这就是为什么我说出来。什么是dermoscopic标准对这些方法得到不同的输出?或者我应该修改代码吗?
*
*我使用黑色素瘤皮肤癌的图像
< <
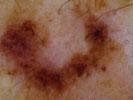
> >
如果真正的
imdata =重塑(grayscaleImage [], 1);
imdata =双(imdata);
[imdx mn] = kmeans (imdata 2);
imIdx =重塑(imdx、大小(replacedImage));
次要情节(2,2,2);
imshow (imIdx []);
轴在;
标题(“k - means方法……”,“字形大小”、字形大小);
imdata1 =重塑(GrayscaleImage [], 1);
imdata =双(imdata);
(imdx mn) = fcm (imdata, 2);
% imIdx =重塑(imdx、大小(replacedImage));
次要情节(2、2、3);
imshow (imIdx []);
轴在;
标题(“模糊c均值方法……”,“字形大小”、字形大小);
结束
0评论
答案(1)
图像分析
2017年7月30日
首先,
imdata =重塑(grayscaleImage [], 1);
imdata =双(imdata);
可以用(:)操作更简单:
imdata =双(grayscaleImage (:));
接下来,你使用kmeans和fcm本质上是相同的,你要想出一个阈值将图像分为前景和背景。这就是imbinarize ()。如果图像有两个明显,分离模式直方图,然后所有3方法很可能决定相同的阈值和给你相同的前景和背景类。如果阈值略有不同,那么他们可能会给你非常相似,但不完全相同的图像。这就是为什么这两种方法你试过本质上是没有区别。
接下来,没有保证自动阈值将会强劲。例如,如果你的图像是完全正常的皮肤吗?你期望它说没有皮肤癌。但是你的自动阈值方法
被强迫
将图像分成两个类,你假设一个类是癌症。为了避免你不得不说,如果两个类是关闭的方式,然后基本上只有一个类,而不是两个。然后你必须决定如果这一形象是100%的正常或100%的癌症。如果你要求你的视野至少有一半的像素是正常的,如果只有一个类,你可以假定它是正常的,不是癌。
