基于深度学习的三维脑肿瘤分割
这个例子展示了如何训练一个三维U-Net神经网络和执行语义分割脑肿瘤从三维医学图像。该示例演示了如何训练一个三维U-Net网络,并提供了一个预训练网络。强烈建议使用支持cuda的NVIDIA™GPU,计算能力3.0或更高,用于3-D语义分割(需要并行计算工具箱™)。
介绍
语义分割包括用类标记图像中的每个像素或三维体素。这个例子说明了如何使用深度学习方法在磁共振成像(MRI)扫描中对脑肿瘤进行二元语义分割。在这种二值分割中,每个像素都被标记为肿瘤或背景。
此示例使用3-D U-Net架构执行脑肿瘤分割[1.]U-Net是一种快速、高效和简单的网络,在语义分割领域已经非常流行。
医学图像分割的一个挑战是存储和处理3d体积所需的内存量。由于GPU资源的限制,在全输入量上训练网络是不切实际的。这个例子通过在图像补丁上训练网络来解决这个问题。该示例使用重叠策略将测试补丁缝合到一个完整的分段测试卷中。该示例通过使用神经网络中卷积的有效部分来避免边界伪影[5.].
医学图像分割的第二个挑战是数据中的类不平衡,当使用传统的交叉熵损失时,会妨碍训练。此示例通过使用加权多类骰子损失函数来解决此问题[4.].对类进行加权有助于抵消较大区域对Dice分数的影响,使网络更容易学习如何分割较小的区域。
下载培训、验证和测试数据
本例使用BraTS数据集[2.].BraTS数据集包含脑肿瘤的MRI扫描,即神经胶质瘤,这是最常见的原发性脑恶性肿瘤。数据文件大小为~ 7gb。如果您不想下载BraTS数据集,那么直接到下载预训练网络和样本测试集节。
创建一个目录来存储BraTS数据集。
imageDir=fullfile(tempdir,“小鬼”);如果~exist(imageDir,“dir”)mkdir(imageDir);结束
要下载BraTS数据,请转到医学分割十项全能网站并点击“下载数据”链接。下载“Task01_.tar”文件[3.].将TAR文件解压到指定的目录中imageDir变量。当成功解压缩,imageDir将包含一个名为Task01_BrainTumour它有三个子目录:imagesTr,影像学家,labelsTr.
该数据集包含750个4-D卷,每个卷代表一堆3-D图像。每个4-D卷的尺寸为240 × 240 × 155 × 4,其中前三个维度对应于一个3-D体积图像的高度、宽度和深度。第四维对应不同的扫描方式。该数据集被分为484个带有体素标签的训练卷和266个测试卷,测试卷没有标签,所以本例没有使用测试数据。相反,该示例将484个训练卷分成三个独立的集,用于训练、验证和测试。
预处理培训和验证数据
为了更有效地训练三维U-Net网络,利用辅助功能对MRI数据进行预处理预处理数据集.这个函数作为支持文件附加到示例中。万博1manbetx
helper函数执行以下操作:
将数据裁剪到一个主要包含大脑和肿瘤的区域。裁剪数据减少了数据的大小,同时保留了每个MRI体积的最关键部分及其相应的标签。
通过减去切割大脑区域的平均值并除以标准偏差,独立地对每个体积的每个形态进行标准化。
将484个训练卷分解为400个训练、29个验证和55个测试集。
预处理数据大约需要30分钟才能完成。
sourceDataLoc=[imageDir filesep“Task01_BrainTumour”];preprocessDataLoc=fullfile(tempdir,“小鬼”,“预处理数据集”);预校准数据集(预校准数据LOC,源数据LOC);
创建用于训练和验证的随机补丁提取数据存储
使用随机的补丁提取数据存储将训练数据提供给网络并验证训练进度。该数据存储从地面真实图像和相应的像素标签数据中提取随机斑块。在进行任意大容量的训练时,修补是一种常见的防止内存耗尽的技术。
创建一个图像数据存储来存储3d图像数据。因为mat文件格式是非标准的图像格式,所以必须使用mat文件阅读器来读取图像数据。你可以使用辅助的mat文件读取器,matRead.这个函数作为支持文件附加到示例中。万博1manbetx
volReader=@(x)matRead(x);volLoc=fullfile(preprocessDataLoc,“imagesTr”);volds = imageDatastore (volLoc,...“文件扩展名”,“.mat”,“ReadFcn”, volReader);
创建一个像素标签数据库存储标签。
lblLoc=fullfile(预处理数据LOC,“labelsTr”);一会= [“背景”,“肿瘤”];pixelLabelID=[0 1];pxds=pixelLabelDatastore(lblLoc,类名,pixelLabelID,...“文件扩展名”,“.mat”,“ReadFcn”, volReader);
预览一个图像卷和标签。显示标记卷使用labelvolshow功能。通过设置背景标签的可见性,使背景完全透明(1.)0.
体积=预览(volds);标签=预览(pxds);viewPnl = uipanel(图,“头衔”,“标记的训练量”);hPred = labelvolshow(标签、体积(:,:,:1),“父”viewPnl,...“LabelColor”,[0 0 0;1 0 0]);hPred.LabelVisibility (1) = 0;
创建一个randomPatchExtractionDatastore包含训练图像和像素标签数据的。指定132×132×132体素的面片大小。指定“PatchesPerImage”在培训期间从每对卷和标签中提取16个随机定位的修补程序。指定最小批量大小为8。
patchSize=[132];patchPerImage=16;miniBatchSize=8;patchds=randomPatchExtractionDatastore(volds、pxds、patchSize、,...“PatchesPerImage”, patchPerImage);patchds。MiniBatchSize = MiniBatchSize;
按照相同的步骤创建一个randomPatchExtractionDatastore包含验证图像和像素标签数据的。您可以使用验证数据评估网络是否随着时间的推移而不断学习、拟合不足或拟合过度。
volLocVal = fullfile (preprocessDataLoc,“imagesVal”);voldsVal = imageDatastore (volLocVal,...“文件扩展名”,“.mat”,“ReadFcn”,volReader);lblLocVal=fullfile(预处理数据loc,“labelsVal”);pixelLabelID pxdsVal = pixelLabelDatastore (lblLocVal,一会,...“文件扩展名”,“.mat”,“ReadFcn”, volReader);dsVal = randomPatchExtractionDatastore (voldsVal pxdsVal patchSize,...“PatchesPerImage”,patchPerImage);dsVal.MiniBatchSize=MiniBatchSize;
方法来增加训练和验证数据变换具有由helper函数指定的自定义预处理操作的函数augmentAndCrop3dPatch.这个函数作为支持文件附加到示例中。万博1manbetx
这个augmentAndCrop3dPatchFunction执行以下操作:
随机旋转和反射训练数据,使训练更具鲁棒性。该函数不旋转或反映验证数据。
作物响应补丁的输出大小的网络,44 × 44 × 44体素。
数据源=“培训”;dsTrain =变换(patchds @ (patchIn) augmentAndCrop3dPatch (patchIn数据源));数据源=“验证”;dsVal =变换(dsVal @ (patchIn) augmentAndCrop3dPatch (patchIn数据源));
设置三维U形网图层
本例使用3-D U-Net网络[1.].在U-Net中,最初的卷积层序列被最大池化层点缀,依次降低了输入图像的分辨率。这些层之后是一系列卷积层,点缀上采样算子,依次增加输入图像的分辨率。在每个ReLU层之前引入了批处理规格化层。U- net这个名字来源于这样一个事实:这个网络可以被画成一个像字母U一样的对称形状。
属性创建默认的3-D U-Net网络unetLayers函数。指定两个类分割。当使用重叠策略预测测试量时,还指定有效的卷积填充以避免边界伪影。
inputPatchSize = [132 132 132 4];numClasses = 2;[lgraph, outPatchSize] = unet3dLayers (inputPatchSize numClasses,“卷积填充”,“有效”);
为了更好地分割较小的肿瘤区域并减少较大背景区域的影响,本例使用dicePixelClassificationLayer.将像素分类层替换为骰子像素分类层。
outputLayer = dicePixelClassificationLayer (“姓名”,“输出”);lgraph = replaceLayer (lgraph,“分割层”,输出层);
数据已在中标准化预处理培训和验证数据这个例子的一部分。数据规范化image3dInputLayer是不必要的,因此将输入层替换为没有数据规范化的输入层。
inputLayer=image3dInputLayer(inputPatchSize,“归一化”,“没有”,“姓名”,“ImageInputLayer”);lgraph = replaceLayer (lgraph,“ImageInputLayer”,输入层);
或者,您也可以使用Deep Learning Toolbox中的Deep network Designer应用程序修改三维U-Net网络™.
绘制更新后的三维U-Net网络图。
analyzeNetwork (lgraph)
指定培训选项
使用亚当优化求解。属性指定超参数设置trainingOptions函数。初始学习率设置为5e-4,并随着训练的持续而逐渐降低。你可以用MiniBatchSize属性,该属性基于您的GPU内存。为了最大限度地提高GPU内存利用率,最好选择大输入补丁而不是大批量。请注意,批量规范化层对于较小的MiniBatchSize.根据初始学习速率调整初始学习速率MiniBatchSize.
选择= trainingOptions (“亚当”,...“MaxEpochs”, 50岁,...“InitialLearnRate”,5e-4,...“LearnRateSchedule”,“分段”,...“LearnRateDropPeriod”,5,...“LearnRateDropFactor”,0.95,...“ValidationData”dsVal,...“ValidationFrequency”, 400,...“情节”,“训练进步”,...“详细”假的,...“MiniBatchSize”,小批量);
下载预训练网络和样本测试集
(可选)从BraTS数据集中下载预训练版本的3-D U-Net和五个样本测试卷及其相应标签[3.].预先训练的模型和样本数据使您能够对测试数据进行分割,而无需下载完整的数据集或等待网络训练。
培训3调谐url=“//www.tianjin-qmedu.com/万博1manbetxsupportfiles/vision/data/brainTumor3DUNetValid.mat”;sampleData_url=“//www.tianjin-qmedu.com/万博1manbetxsupportfiles/vision/data/sampleBraTSTestSetValid.tar.gz”;imageDir=fullfile(tempdir,“小鬼”);如果~exist(imageDir,“dir”)mkdir(imageDir);结束downloadTrained3DUnetSampleData (trained3DUnet_url sampleData_url imageDir);
下载预先训练的3d U-Net数据集。下载需要几分钟…完成了。下载样本BraTS测试数据集。下载和解压需要几分钟。完成了。
列车网络
在配置培训选项和数据源后,使用trainNetwork函数。要训练网络,设置溺爱变量为真正的.具有CUDA功能的NVIDIA™ 强烈建议培训使用计算能力为3.0或更高的GPU。
如果你保留溺爱以下代码中的变量为假的,则该示例返回一个预先训练的3-D U-Net网络。
注意:在4个NVIDIA™Titan Xp GPU的多GPU系统上,培训需要大约30个小时,根据你的GPU硬件,可能需要更长的时间。
doTraining = false;如果doTraining modelDateTime = datestr“dd-mmm-yyy-HH-MM-SS”);(网络,信息)= trainNetwork (dsTrain、lgraph选项);保存([“trained3DUNetValid -”模型日期时间“-时代-”num2str(options.MaxEpochs)“.mat”],“净”);其他的inputPatchSize=[132 4];outPatchSize=[44 2];加载(完整文件)(imageDir,“trained3DUNet”,“brainTumor3DUNetValid.mat”));结束
你现在可以使用U-Net来对脑肿瘤进行语义分割。
对测试数据进行分割
强烈建议使用GPU对图像进行语义分割(需要并行计算工具箱™)。
选择测试数据的来源,其中包含地面真值卷和测试标签。如果你保留useFullTestSet以下代码中的变量为假的,则示例使用5卷进行测试。如果你设置useFullTestSet变量来真正的,则示例使用从完整数据集中选择的55张测试图像。
useFullTestSet=false;如果usfulltestset volLocTest = fullfile(preprocessDataLoc,“imagesTest”);lblLocTest = fullfile (preprocessDataLoc,“labelsTest”);其他的volLocTest = fullfile (imageDir,“sampleBraTSTestSetValid”,“imagesTest”); lblLocTest=fullfile(imageDir,“sampleBraTSTestSetValid”,“labelsTest”);一会= [“背景”,“肿瘤”];pixelLabelID = [0 1];结束
这个voldsTest变量存储地面真值测试图像。这个pxdsTestVariable存储ground真值标签。
volReader=@(x)matRead(x);voldsTest=图像数据存储(volLocTest,...“文件扩展名”,“.mat”,“ReadFcn”, volReader);pixelLabelID pxdsTest = pixelLabelDatastore (lblLocTest,一会,...“文件扩展名”,“.mat”,“ReadFcn”, volReader);
使用重叠策略来预测每个测试量的标签。每个测试体积被填充,使输入大小为网络输出大小的倍数,并补偿有效卷积的影响。重叠算法选择重叠的patch,利用该算法预测每个patch的标签语义词组功能,然后重新组合补丁。
id = 1;而hasdata (voldsTest) disp ([“处理测试量”num2str (id)]);tempGroundTruth =阅读(pxdsTest);groundTruthLabels {id} = tempGroundTruth {1};{id} =阅读卷(voldsTest);%对测试图像使用反射填充。%避免填充不同的模式。volSize =大小({id}卷,(1:3));padSizePre = (inputPatchSize (1:3) -outPatchSize (1:3)) / 2;padSizePost = (inputPatchSize(1:3)-outPatchSize(1:3))/2 + (outPatchSize(1:3)-mod(volSize,outPatchSize(1:3)));volPaddedPre = padarray (padSizePre卷{id},“对称”,“pre”); volPadded=padarray(volPaddedPre、padSizePost、,“对称”,“职位”);[heightPad, widthPad depthPad ~] =大小(volPadded);(高度、宽度、深度、~)=大小(卷{id});tempSeg =分类(0(高度、宽度、深度,“uint8”),[0, 1],类名);%重叠的策略分割的体积。对于k = 1: outPatchSize (3): depthPad-inputPatchSize (3) + 1对于j = 1: outPatchSize (2): widthPad-inputPatchSize (2) + 1对于i = 1:outPatchSize(1):heightPad-inputPatchSize(1)+1 patch = volpads (i:i+inputPatchSize(1)-1,...j: + inputPatchSize (2) 1,...k:k+inputPatchSize(3)-1,:);patchSeg=semanticseg(patch,net);tempSeg(i:i+outPatchSize(1)-1,...j: + outPatchSize (2) 1,...凯西:k + outPatchSize (3) 1) = patchSeg;结束结束结束%裁剪出额外的填充区域。tempSeg = tempSeg(1:身高、1:宽度、1:深度);%保存预测的体积结果。predictedLabels {id} = tempSeg;id = id + 1;结束
处理测试卷1处理测试卷2处理测试卷3处理测试卷4处理测试卷5
比较地面真实与网络预测
选择一幅测试图像来评估语义分割的准确性。从4-D体积数据中提取第一个模态,并将该3-D体积存储在变量中vol3d.
volId = 1;vol3d = {volId}卷(::,:,1);
沿深度方向以蒙太奇方式显示地面真实和预测标签的中心切片。
zID=尺寸(vol3d,3)/2;zSliceGT=labeloverlay(vol3d(:,:,zID),GroundtruthletLabels{volId}(:,:,zID));zSlicePred=labeloverlay(vol3d(:,:,zID),预测标签{volId}(,:,zID));图形蒙太奇({zSliceGT,zSlicePred},“大小”,[1 2],“边界大小”,5)头衔(“标记的地面真相(左)与网络预测(右)”对比)

使用以下命令显示标有“音量”的地面实况labelvolshow功能。通过设置背景标签的可见性,使背景完全透明(1.)0。因为肿瘤位于脑组织内部,所以要使一些脑体素透明,以便肿瘤可见。要使一些脑体素透明,请将体积阈值指定为[0,1]范围内的数字。所有低于此阈值的标准化体积强度都是完全透明的。此示例将体积阈值设置为小于1,以便某些大脑像素保持可见,从而为肿瘤在大脑中的空间位置提供上下文。
viewPnlTruth=uipanel(图,“头衔”,“地面真相标记卷”);hTruth = labelvolshow (groundTruthLabels {volId}, vol3d,“父”,viewPnlTruth,...“LabelColor”,[0 0 0;1 0 0],“VolumeThreshold”, 0.68);hTruth.LabelVisibility (1) = 0;
对于相同的卷,显示预测的标签。
viewPnlPred=uipanel(图,“头衔”,“预测标签卷”);hPred = labelvolshow (predictedLabels {volId}, vol3d,“父”,viewPnlPred,...“LabelColor”,[0 0 0;1 0 0],“VolumeThreshold”, 0.68);
hPred.LabelVisibility (1) = 0;

此图显示了在其中一个卷上按顺序显示切片的结果。标记的地面真相在左侧,网络预测在右侧。

量化细分精度
使用掷骰子函数。该函数计算预测和基本真值分割之间的骰子相似系数。
diceResult=0(长度(voldsTest.Files),2);对于j = 1:length(vol) dicerresult (j,:) = dice(groundTruthLabels{j},predictedLabels{j});结束
计算测试卷集合中的平均骰子分数。
meansdicebackground=平均值(diceResult(:,1));disp([“背景的平均骰子分数”num2str (j),...‘测试卷’num2str (meanDiceBackground)])
5个测试卷的平均骰子得分= 0.9993
meanDiceTumor =意味着(diceResult (:, 2));disp ([“肿瘤的平均骰子得分”num2str (j),...‘测试卷’,num2str(平均肿瘤)])
5个试验体积肿瘤的平均骰子得分= 0.9585
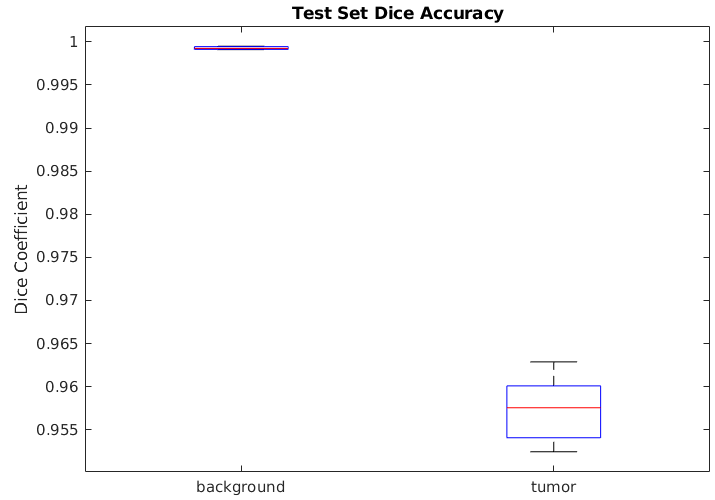
图中显示了一个箱线图它可视化了5个样本测试卷中Dice分数的统计数据。图中的红线表示类的骰子值的中值。蓝色框的上下限分别表示第25和75个百分点。黑须延伸到最极端的数据点,而不考虑异常值。

如果你有统计和机器学习工具箱™, 然后你可以使用箱线图函数用于可视化所有测试卷中骰子分数的统计信息。创建箱线图,设置createBoxplot变量为真正的.
createBoxplot = false;如果createBoxplot图boxplot(结果)标题(“测试集骰子精度”)Xticklabel(类名)ylabel(“骰子系数”)结束
工具书类
[1] çiçek,Ö,A.Abdulkadir,S.S.Lienkamp,T.Brox和O.Ronneberger.“3D U-Net:从稀疏注释学习密集体积分割”,摘自国际医学图像计算和计算机辅助干预会议论文集- MICCAI 2016.希腊雅典,2016年10月,第424-432页。
Isensee, F., P. Kickingereder, W. Wick, M. Bendszus, K. H. Maier-Hein。脑肿瘤分割和放射组学生存预测:对BRATS 2017挑战的贡献在国际MICCAI脑病研讨会论文集加拿大魁北克市,2017年9月,第287-297页。
[3] “脑瘤”。十项全能。http://medicaldecathlon.com/
BraTS数据集由医学细分十项全能提供CC-BY-SA 4.0许可证。拒绝所有保证和声明;详细信息请参见license。属性中链接的数据集已被MathWorks®修改下载预训练网络和样本测试集修改后的样本数据集被裁剪到一个主要包含大脑和肿瘤的区域,每个通道通过减去平均值并除以裁剪后的大脑区域的标准偏差进行独立标准化。
苏德雷,c.h., W. Li, T. Vercauteren, S. Ourselin, M. J. Cardoso。“广义骰子重叠作为高度不平衡分割的深度学习损失函数。”医学图像分析中的深度学习和临床决策支持的多模态学习:第三届国际研讨会万博1manbetx.加拿大魁北克市,2017年9月,第240-248页。
Ronneberger, O., P. Fischer和T. Brox。U-Net:用于生物医学图像分割的卷积网络。在国际医学图像计算与计算机辅助干预会议论文集- MICCAI 2015德国慕尼黑,2015年10月,第234-241页。可从arXiv获得:1505.04597。
另见
dicePixelClassificationLayer|图像数据存储|像素标签数据库|randomPatchExtractionDatastore|语义词组|trainNetwork|trainingOptions|变换

