使用Genomics Viewer应用程序可视化NGS数据
该基因组浏览器应用程序允许您查看和与综合基因组浏览器的嵌入式版本集成探索基因组数据(IGV)[1][2]。基因组数据包括NGS读取比对,基因组的变体,和分段拷贝数数据。
本例的第一部分简要介绍了应用程序和所支持的文件格式。万博1manbetx这个例子的第二部分探索了细胞色素p450基因(CYP2C19)中的单个核苷酸变异。
打开应用程序
在命令行键入genomicsViewer。或者,点击应用程序图标应用选项卡。该应用程序需要互联网连接。
默认情况下,应用程序加载人(GRCh38 / hg38)作为参考序列和基因REFSEQ作为注释文件。有在应用两个主面板。左侧面板是跟踪面板,右边的面板是嵌入的IGV Web应用程序。该跟踪面板是只读区域中显示曲目名称,源文件名,并跟踪类型。该跟踪面板更新相应的配置在嵌入式应用IGV的轨道。
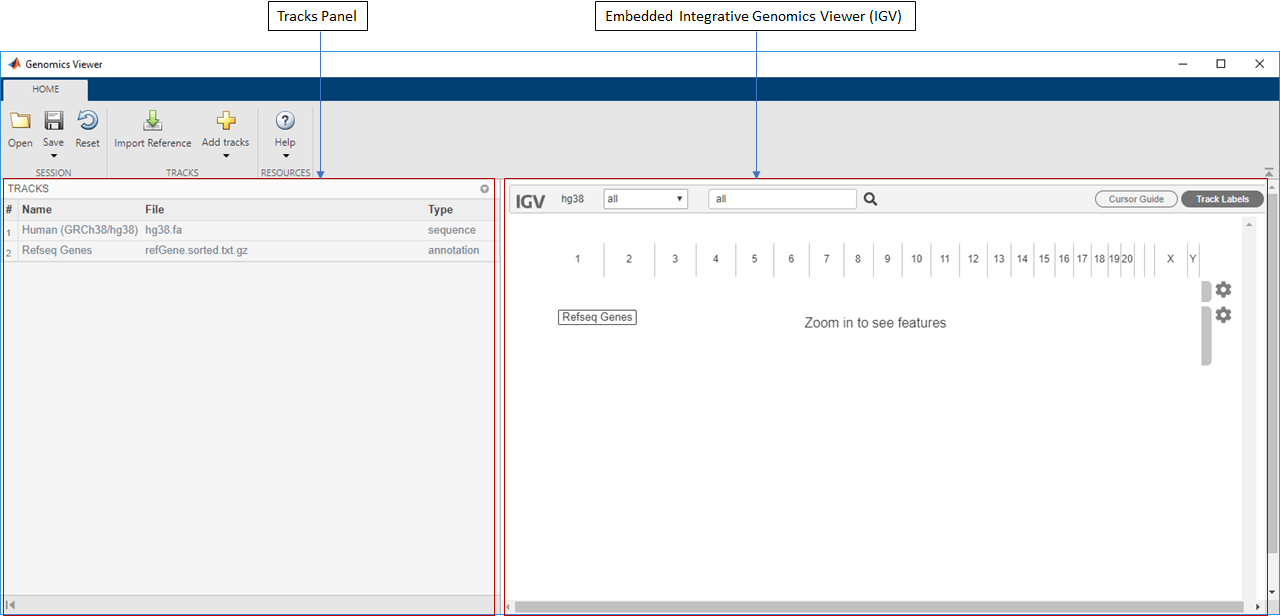
该重置按钮将应用程序恢复到带有两个轨道的默认视图(带有Refseq基因的HG38),并删除任何其他现有的轨道。在重置之前,你可以将当前视图保存为会话(以.json)文件,并在以后恢复它。
通过导入数据添加轨迹
进口参考序列
您可以导入一个引用序列。引用序列必须在FASTA文件中。选择导入参考在家选项卡。您也可以导入包含细胞遗传学G显带数据对应的染色体区段文件。您可以添加本地文件或指定外部URL。该URL必须以启动HTTPS或GS。其他文件传输协议,如ftp,不支持。万博1manbetx
导入序列读取对齐数据
序列读取校准数据可以导入多个数据集。对齐数据必须是BAM或CRAM文件。它不要求你有相应的索引文件(.BAI或.CRAI)在同一位置为您的BAM或CRAM文件。然而,如果没有索引文件会使应用程序变慢。
您可以添加读取对齐文件使用从文件添加曲目和从URL添加曲目从选项添加跟踪按钮。如果指定URL, URL必须以任意一个开头HTTPS或GS。其他文件传输协议,如ftp,不支持。万博1manbetx
导入功能注释和其它基因组数据
您可以从包含单个引用序列数据的多个文件中导入多组特性注释。支持的注万博1manbetx释文件是:请全部,.GFF,.GFF3,.GTF。
您也可以导入结构变异(.VCF)和可视化的遗传改变,如插入和缺失。
你可以查看分段副本编号数据(.SEG)和定量基因组数据(。假发,.BIGWIG,.BEDGRAPH),如芯片峰和对准覆盖。
您可以通过添加注释和基因组数据文件从文件添加曲目和从URL添加曲目从选项添加跟踪按钮。如果指定URL, URL必须以任意一个开头HTTPS或GS。其他文件传输协议,如FTP,不支持。万博1manbetx
观察细胞色素P450中单核苷酸的变化
该CYP2C19该基因属于细胞色素P450基因家族。由细胞色素P450基因产生的酶参与细胞内各种分子和化学物质的代谢。CYP2C19酶在至少10%的常用药物代谢中起作用[3]。细胞色素p450家族的多态性可能在个体中引起药物不良反应。单核苷酸变异的一个例子是rs4986893在位置CHR 10:94780653哪里G被替换一种。这种等位基因变体也被称为CYP2C19 * 3。下面的步骤展示了如何使用低覆盖率和高覆盖率数据在应用程序中可视化这些变化。
加载会话文件
出于本例的目的,我们从一个预加载了一些轨道的会话文件开始。要加载文件,请单击打开。导航matlabroot\ \ bioinfo \例子,其中matlabroot这个文件夹是您安装MATLAB的地方吗®。选择rs4986893.json。
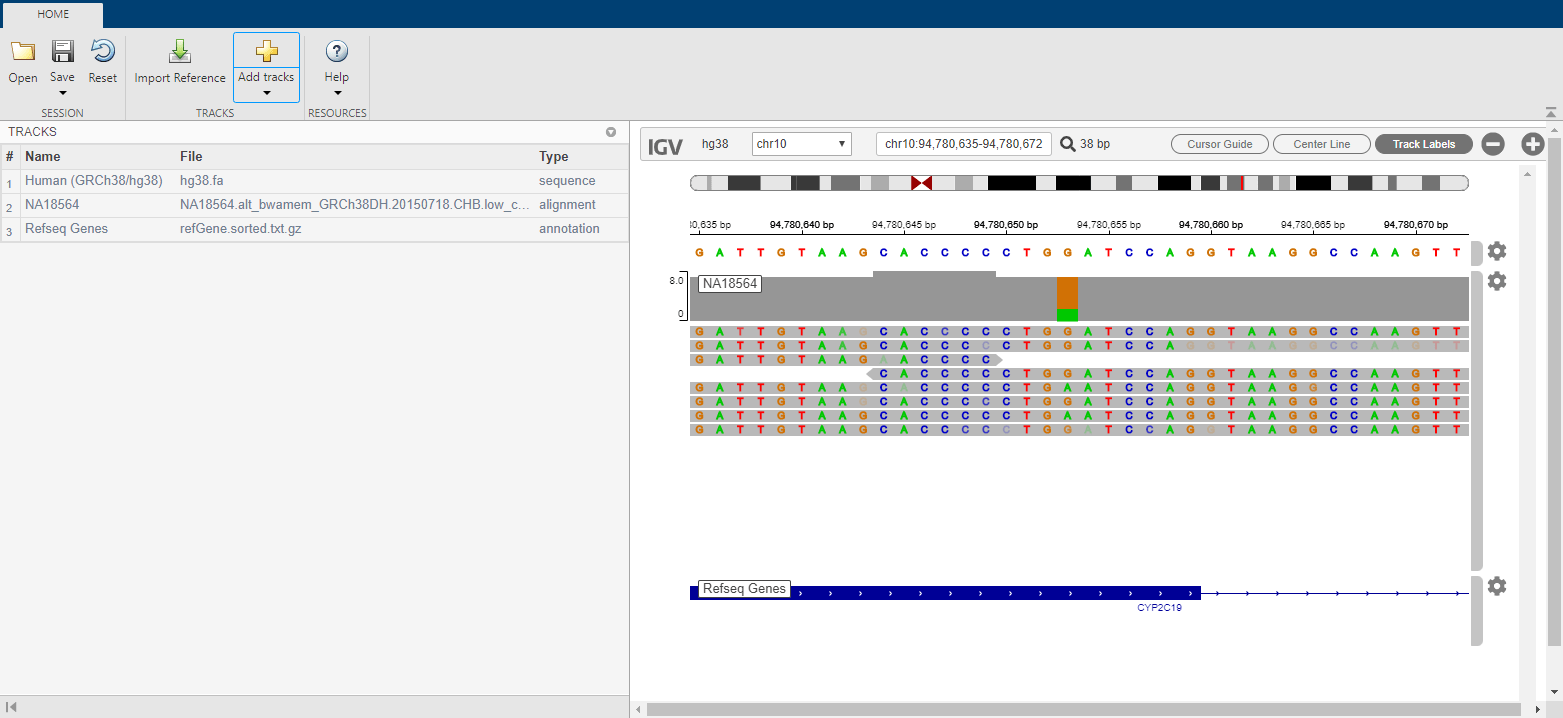
会议包含三个轨道:
人类(GRCh38 / hg38)作为一个参考
NA18564如低覆盖率对齐数据
以RefSeq基因
低覆盖率对齐数据来自女性中国汉族来自北京,中国。样本ID是NA18564样品已经被鉴定为CYP2C19 * 3突变[4]。
探索低覆盖率数据
在本次会议文件,校准数据已经围绕着对突变的位置CYP2C19基因。
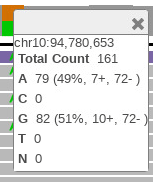
单击覆盖区域的橙色条看位置和等位基因分布信息。
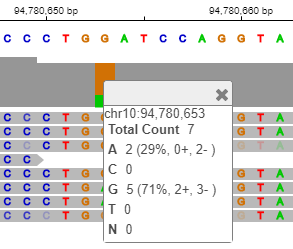
它表明的71%读取有g,而29%具有在该位置处CHR 10:94780653。这个数据是覆盖率低的数据可能不会显示该变异的所有出现。高覆盖率的数据将在后面的实例探讨。
关闭数据提示窗口。
您可以自定义应用程序中的数据显示的各个方面。例如,你可以改变轨道高度,使供以后轨道更多的空间。单击第二个齿轮图标。选择
设置轨道高度。输入200。
有关嵌入式IGV应用程序及其可用选项的详细信息,请访问在这里。
探索高覆盖率数据
你可以看一下从同一样品的高覆盖率的数据看到这个突变的发生。
去国际基因组样本资源网站。
搜索样本NA18564。
下载外显子组对齐文件,它是在
.CRAM格式。目录中相应的索引文件
.CRAI格式。该文件在同一位置保存为源.CRAM文件。按钮上的(+)图标家选项卡。选择下载
.CRAM文件并点击打开。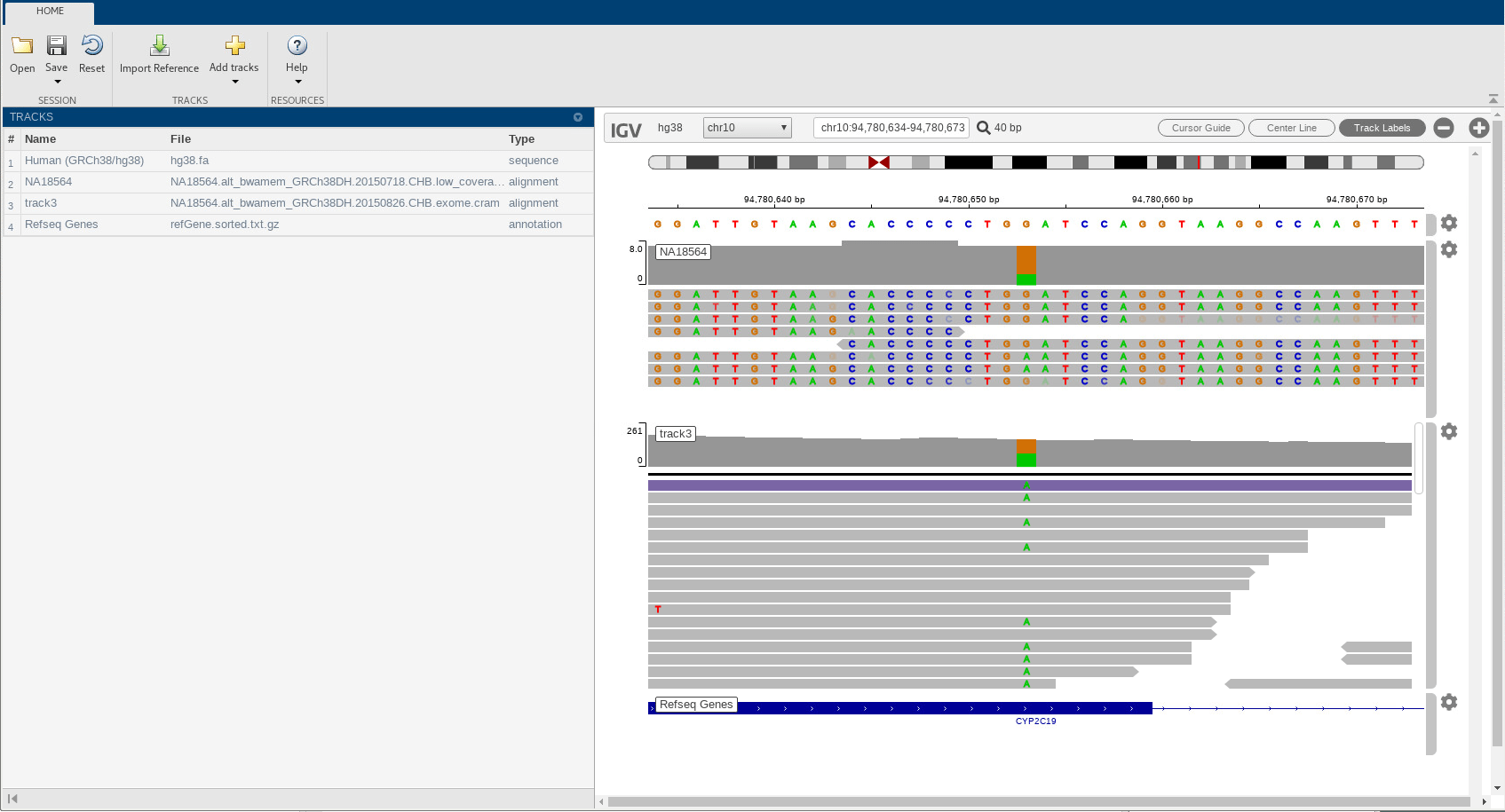
高覆盖率数据显示为track3。您现在可以在多次读取中看到该突变的多次出现。
点击覆盖区域的橙色条可以看到等位基因的分布。它表明,G被A取代的几率接近50%。
参考文献
[1] Robinson, J. Thorvaldsdottir, W. Winckler, M. Guttman, E. Lander, G. Getz, J. Mesirov. 2011。结合基因组学查看器。自然生物技术。29:24-26。
Thorvaldsdottir, H., J. Robinson, J. Mesirov, 2013。综合基因组查看器(IGV):高性能基因组数据可视化和探索。生物信息学简报。14:178-192。





