使用SimBiology模型构建器将SGLT2抑制纳入基于生理的葡萄糖-胰岛素模型
这个例子展示了如何使用一种假设的化合物将SGLT2抑制添加到现有的葡萄糖-胰岛素模型中Simbiology Model Builder..
葡萄糖 - 胰岛素模型
该模型是葡萄糖 - 胰岛素模型的另一个偶像实现模拟葡萄糖 - 胰岛素反应的例子。该模型是基于Dalla Man等人发表的论文。在2007年的出版物中[1],作者开发了一个人餐后葡萄糖-胰岛素反应模型。该模型使用常微分方程描述系统的动力学。作者使用他们的模型来模拟正常人和有各种胰岛素损伤的人在一餐或多餐后的葡萄糖-胰岛素反应。
钠-葡萄糖协同转运体-2 (SGLT2)抑制
已显示SGLT2受体促进约50%的肾葡萄糖重吸收[3].该示例假定具有假设的SGLT2抑制剂化合物,其抑制SGLT2达50%。还假设合理的给药方案和PK属性。在该实施例中,您将该抑制剂化合物的药代动力学/药效学(PK / PD)包含到葡萄糖 - 胰岛素模型中。
通过添加和配置反应加入抑制剂PK
在以下步骤中,您通过使用两种反应来模拟假假假设的SGLT2抑制剂化合物的复合吸收和间隙。
负荷模型
打开Simbiology Model Builder.通过点击查看Simbiology Model Builder.在这一点应用标签或打字
SimbiologyModelBuilder.在命令行。在这一点家应用程序的选项卡,选择打开.
导航到文件夹
matlabroot.\ \ simbio \ \数据示例.matlabroot.是您已安装MATLAB的文件夹。进入matlabroot.在命令行,为您提供了根本文件夹的路径。选择命名的项目文件sglt2_model_incomplete.sbproj..点击打开.
添加和配置反应
笔记
在麦克斯,使用命令钥匙而不是Ctrl..
从工具栏中拖放两个物种块图表标签。
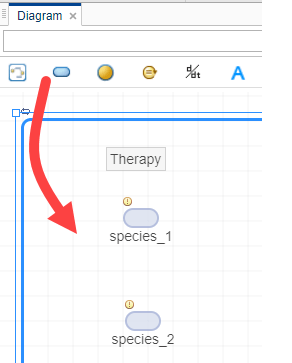
按Ctrl.从第一个物种拖一条线到第二个物种。一个反应块出现在中间。这个反应表示化合物的吸收。
通过双击它来编辑默认物种名称。改名
species_1到gi_sglt2_inhib.和species_2到plasma_sglt2_inhib..
单击反应块。在里面属性编辑器右边的窗格,改变反应名称到
复合_吸附.在里面动力学法部分,改变自动理解向前速率参数克到
k_compound_absorption..该模型已经具有前向率参数k_compound_absorption.这是之前创建的。该应用程序在反应速率表达式中使用绿色文本进行参数名称和蓝色文本的物种名称。小费
要更改默认反应配置,请单击喜好在这一点家标签。在“首选项”对话框中,单击建筑模型.在里面反应建设部分,有三个选项来更改默认的动态法,为动态法创建参数,并更改创建参数的范围。
在里面状态表,设置两个物种的单位
毫克.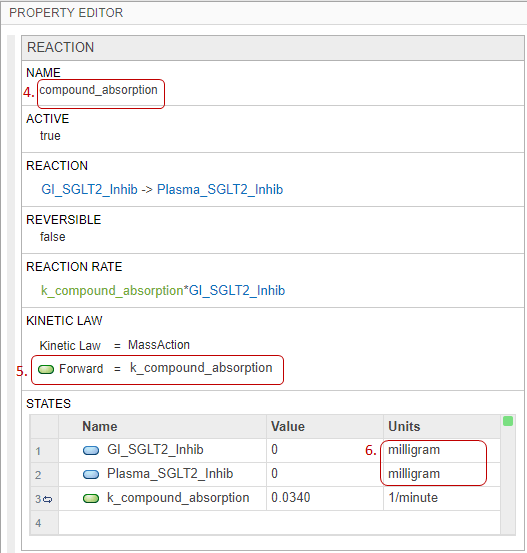
从图表工具栏中拖放另一个反应块以模拟复合间隙。
按Ctrl.并拖动一条线
plasma_sglt2_inhib.反应。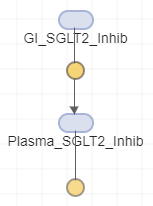
小费
如果块没有对齐,您可以使用对齐工具对齐它们。在这一点家选项卡,选择图工具>
图对齐工具.单击反应块。在里面属性编辑器窗格,改变反应名称到
复合_污垢.更新反应速度到
CL / VD * PLASMA_SGLT2_INIB.CL.和vd.分别表示分布间隙和分布量的模型参数。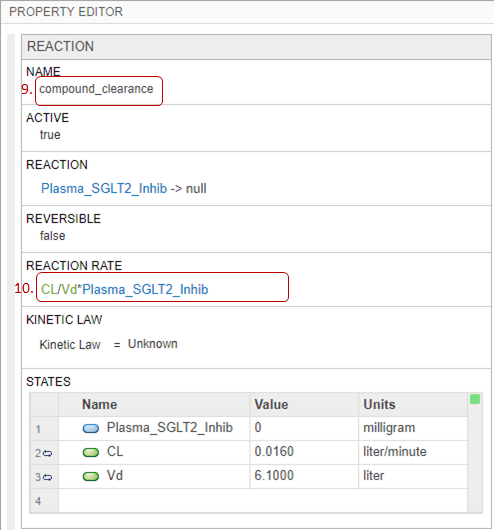
小费
新加入反应的动力学定律是这样的mass默认情况下,和Simbiology Model Builder.应用程序自动创建并映射反应速率所需的物种和参数。对于其他动态法律,仅创建和映射参数。您需要手动创建和映射物种。使用未知动力学定律用自己的参数定义自定义反应速率。您必须定义并添加自定义速率所需的物种和参数。
这mass和未知即使当反应速率是相同的,动力学定律也可以具有不同的模拟结果。当您在不同隔间中的物种具有可逆反应时,可能会发生这种情况。仿真结果的差异是因为在尺寸分析期间通过辛生物进行的体积缩放。有关详细信息,请参阅从反应中获得杂散.具体而言,对于mass,SimBiology使用相应的隔间卷来乘以前向和反向速率。但是,对于未知和其他内置的动力学定律,SimBiology只将整个速率乘以一个包含反应物的隔间。要确切地了解用于缩放的隔间容量,请打开方程式标签并检查杂散部分。
使用数学方程掺入抑制剂PD
SimBiology允许您定义数学表达式以在仿真期间定义或更新型号的值。有关详细信息,请参阅素质模型规则的定义与评估.在以下步骤中,添加重复的分配规则以通过定义基于复合功效排出血浆葡萄糖的肾阈值来掺入抑制剂药效学。
在里面浏览器窗格,单击浏览器工具栏上的加号和选择
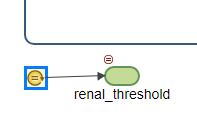
添加重复的分配.该应用程序将焦点移动到最后一个空行中重复分配桌子。双击该行并输入以下表达式,该表达式表示基于Hill方程的复合抑制:
renal_threshold = basal_renal_threshold *(1-compound_imax * plasma_sglt2_inhib ^ 2 /(compound_ic50 ^ 2 + plasma_sglt2_inhib ^ 2))
这图表标签现在显示了重复的分配规则块
renal_threshold.范围。小费
通过它查看整个模型和平移,展开模型评估工具在浏览器窗格并点击概述.
笔记
该应用程序只显示一个参数块,用于重复分配规则、速率规则或事件函数的左侧参数(LHS)。
该应用程序仅显示重复分配和速率规则的规则块。
该应用程序使用虚线连接规则右侧的数量。默认情况下,不显示这些行。要显示这些行,请单击规则块。从属性编辑器窗格中,在块部分,集表达线到显示.
更新肾脏排泄反应,以掺入抑制剂化合物的存在
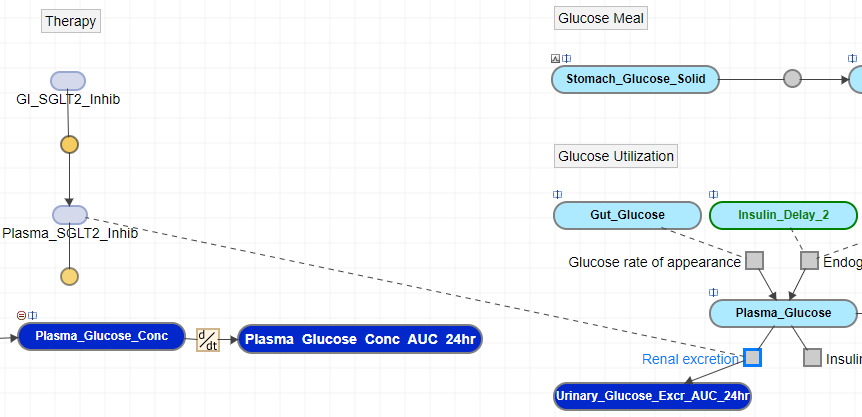
该模型的肾脏排泄反应目前被定义为Plasma_Glucose - > Urinary_Glucose_Excr_AUC使用反应率参数glucose_excretion..速率参数由重复的分配规则定义为glucose_excretion =(plasma_glucose> renal_threshold)* gfr *(plasma_glucose-renal_threshold), 在哪里肾小球滤过率(GFR)是一种决定反应的通量的肾小球过滤速率,并对SGLT2抑制效果产生影响。
在以下步骤中,您更新肾脏排泄反应plasma_glucose + plasma_sglt2_inhib - > plasma_sglt2_inhib + urinary_glucose_excr_auc,其中抑制剂化合物plasma_sglt2_inhib.既是反应的反应物和产物。
在里面图表选项卡,单击命名的灰色方形反应块
肾脏排泄.
在里面属性编辑器窗格,更新反应字符串到
plasma_glucose + plasma_sglt2_inhib - > plasma_sglt2_inhib + urinary_glucose_excr_auc.立即连接线路plasma_sglt2_inhib.对反应块图表标签。笔记
Simbiology使用虚线表示物种既是反应的反应物和产物,也不被反应消耗。
分裂和克隆块
当有多个引用相同的数量时,多行连接到块。要使图表更清晰,可以拆分块,即创建同一块的副本,以便每个引用连接到块的其他副本。您还可以克隆一个块以添加另一个块。例如,您可以首先将物种块克隆到多个表达式中引用。然后,您可以在构建模型时在每个表达式中使用每个克隆。
在以下步骤中,您克隆了plasma_sglt2_inhib.块。这些步骤是可选的,对模型行为没有任何影响。
单击
plasma_sglt2_inhib.图中的块。在里面属性编辑器窗格,滚动到分裂部分。
点击克隆.

在里面图表选项卡时,克隆块出现在原始块的旁边。现在每个块都有一个克隆指示器。
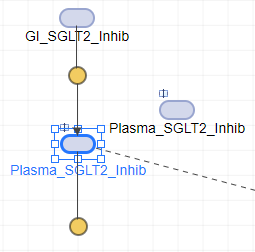
您现在可以将虚线移动到克隆块。首先单击虚线。按Ctrl.并将虚线行拖动到克隆块。当线靠近克隆块时,会出现绿色加号。释放鼠标以将行附加到克隆块。
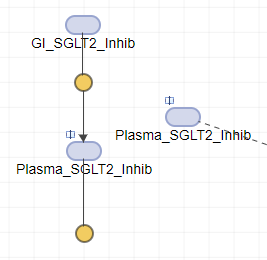
您现在可以将克隆块更近地移动到
肾脏排泄反应块使图更易于阅读。
使用事件纳入模型行为的突然变化
您可以根据指定条件模拟模型行为的突然变化。例如,您可以在特定时间点或交叉某个浓度阈值时重置物种量。SimBiology允许您使用称为事件的建模组件来模拟此类更改。一个事件允许您在自定义条件变为真的时指定在发生的数量值中的离散转换。这种情况称为事件触发器。一旦条件变为真,执行一个或多个事件功能。有关详细信息,请参阅素质模型中的事件.
在以下步骤中,通过添加一个事件触发器和三个事件功能,每24小时将尿葡萄糖总量重置为零。
单击浏览器工具栏上的加号图标。选择
添加事件.该应用程序将焦点移动到最后一个空行中活动桌子。输入以下事件触发器:
地板(时间/蒂米定)> num_day.下一个eventfcn.行,进入
urinary_glucose_excr_auc = 0..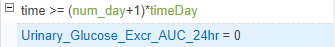
要在同一事件中添加第二个事件函数,请转到属性编辑器事件的窗格。在里面活动FCNS.表,双击空行并输入以下内容以重置天数:
num_day =楼层(时间/ 1440).添加第三个事件功能,通过输入来重置总血糖浓度
plasma_glucose_concentration_auc = 0..
添加剂量
SimBiology让您模拟了由于刺激或药物口服或静脉内施用的刺激物种的增加。为了模拟物种量的增加,使用剂量建模组件。在以下步骤中,您可以模拟抑制剂药物的摄入量,例如每天一次X通过给药时的天数gi_sglt2_inhib.物种。
在里面浏览器窗格中,单击显示剂量工具栏上的图标。
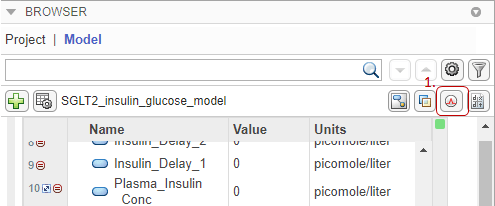
这剂量选项卡出现了。在里面剂量部分,每行代表一剂。这类型列允许您选择重复剂量(默认)和安排剂量.这活跃的列允许您在模拟模型时选择要应用的剂量。
双击名称列在最后一个空行中并输入
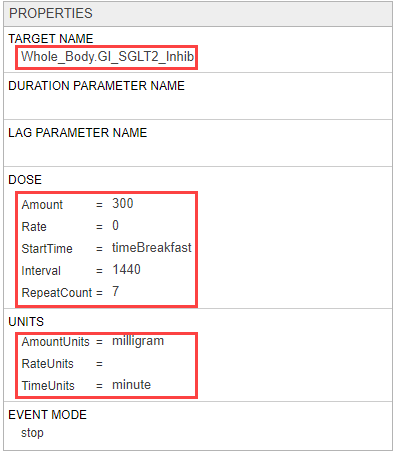
SGLT2抑制QD.在里面特性部分,for.目标名称, 进入
gi_sglt2_inhib.并选择全_body.gi_sglt2_inhib..在里面剂量部分,输入以下内容:
数量=
300速度=
0.开始时间=
60.间隔=
1440重复=
7.
在里面单位部分,输入以下内容:
AmountUnits=
毫克速度=
时刻=
分钟
代表使用变体的生物变异性
您可以使用一个称为的建模组件来建模生物的可变性变体.变型是具有替代值的数量集合。例如,在该示例中,您可以对类型2糖尿病患者的一组参数值和没有类型2糖尿病的患者的另一组值。
出于此示例的目的,模型已具有两个变体。在以下步骤中,您打开了变体选项卡,您可以在其中编辑或添加更多变体。
在里面浏览器窗格中,单击显示变体工具栏上的图标。
这变体选项卡出现了。在里面变体部分,每行代表一个变体。这活跃的列允许您在模拟模型时选择要应用的变体。您可以选择多个变体,如果有数量值的重复规范,则在仿真期间使用变体阵列中的值的最后一次发生。该应用程序在从上到下的表格中显示的顺序应用变体。重新排序变量可以改变初始条件,因为变体以新顺序应用。确保在模拟模型时提供正确的顺序模型分析应用程序。这价值列在内容在应用所选的所有变体后,部分显示最终数量值。
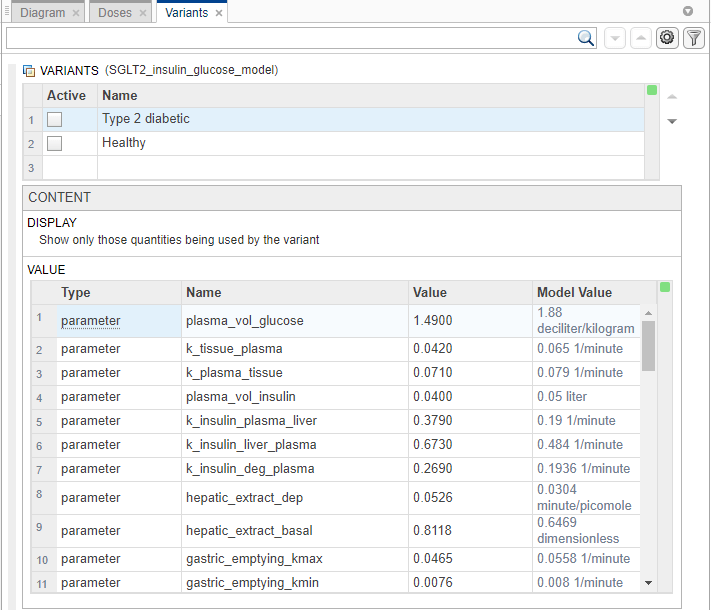
默认情况下,内容部分仅显示了变体修改的数量。要查看所有型号数量,请选择
显示模型中的所有数量在展示部分。
显示模型方程和初始条件
您可以查看底层的等式系统,即表示模型的常规方程(杂志)和规则。SimBiology源于模型反应中的杂散,并且ODES定义了在模型仿真期间集成的数量。有关详细信息,请参阅模型仿真.
您可以使用模型方程和初始条件来调试模型。例如,可以检查ode的初始条件,以查看数量值是否按预期初始化。您还可以看到SimBiology是如何通过用隔间体积除以等式右边来修正ode的尺寸的。容量校正信息可以帮助您调试意外的模拟结果,特别是当您拥有具有不同容量的多室模型时。
要查看模型方程,请单击显示模型方程式工具栏上的图标浏览器窗格。
![]()
该应用程序打开了方程式标签。
反应通量
默认情况下,应用程序在模型方程中显示时嵌入反应通量。清除嵌入势态复选框,以查看助丝部分分开。
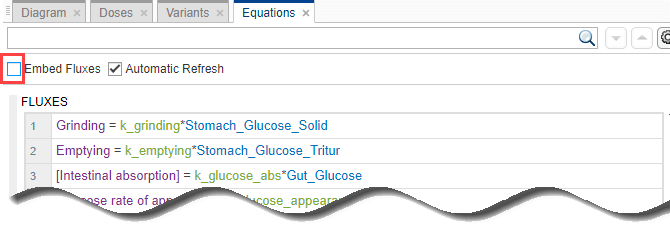
通常,反应助熔剂等同于反应速率,不同之处在于势量尺寸始终是金额/时间.反应速率的尺寸可以在浓度/时间要么金额/时间.有关详细信息,请参阅从反应中获得杂散.
初始条件
您可以查看模型数量的初始条件,即隔间,种类和参数。初始条件是模拟时间的数量值= 0.在工具栏上浏览器窗格,选择查看模型文档选项>显示型号初始条件.
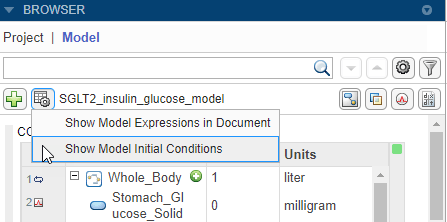
该应用程序添加了一个名为的列初始条件到了隔间和物种表和参数桌子。

定义可观察表达式
SimBiology允许您通过定义和评估自定义表达式来执行后期计算计算。这样的表达被称为可观察到的.在以下步骤中,您将可观察到的表达添加到模型中以计算等离子体葡萄糖的浓度 - 时间轮廓的Cmax和平均值。
单击浏览器工具栏上的加号图标。选择
添加可观察到的.该应用程序将焦点移动到最后一个空行中可观察到桌子。双击空行并输入以下表达式以获取CMAX值:
cmax_plasma_glucose = max(plasma_glucose_concentation).双击下一个空行并输入以下表达式以获取平均值:
Mean_plasma_glucose =均值(plasma_glucose_concentation).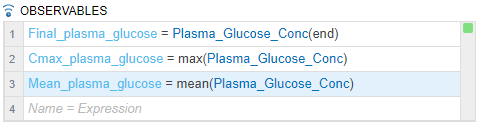
小费
要查看可观察表达式的值,请使用该模拟模型Simbiology Model Analyzer应用程序。跑过模拟模型与之诉讼后处理:计算可观察到的一步。在里面LastRun程序模拟结果的文件夹,展开结果.向载体值的可观察到列出结果.标量值可观察到,例如cmax.和的意思是在本例中,列在名为标量.您可以将数据拖放到数据表或绘图。有关更多型号分析示例,请参阅Simbiology Model Analyzer.
出口模式
Simbiology Model Builder.允许您将模型导出为各种文件格式。你可以:
将模型导出到MATLAB工作区。一旦模型在工作区中,您就可以以编程方式工作。有关更多命令行示例,请参阅构建模型.
将模型导出到SBML文件。
将模型导出到HTML文件。
将模型方程导出到HTML文件中。
将模型组件导出为Excel®文件。
在这一点家标签,在模型部分,选择出口.

小费
您可以在中找到此示例的完整模型matlabroot.\ \ simbio \ data \ SGLT2_model.sbproj例子, 在哪里matlabroot.是您安装MATLAB的文件夹。
参考文献
[1]Dalla Man, Chiara, Robert A. Rizza和Claudio Cobelli。《葡萄糖-胰岛素系统的膳食模拟模型》生物医学工程的IEEE交易54岁的没有。10(2007年10月):1740-49。https://ieeexplore.ieee.org/document/4303268..
[2]达拉姆,Chiara,M. Camilleri和C. Cobelli。“口腔葡萄糖吸收的系统模型:金标准数据验证。”生物医学工程的IEEE交易53岁的没有。12(2006年12月):2472-78。https://ieeexplore.ieee.org/document/4015600..
[3]怀特、欧内斯特·M、唐纳德·d·f·卢和布鲁斯·平山。《人钠葡萄糖转运体的生物学》。生理评论91,没有。2(2011年4月):733-94。https://journals.physiology.org/doi/full/10.1152/physrev.00055.2009.
也可以看看
Simbiology Model Analyzer|Simbiology Model Builder.